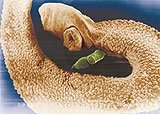
VANDERLEI RODRIGUES / USPCasal: durante a reprodução, a fêmea do Schistosoma (verde) se instala dentro do macho.VANDERLEI RODRIGUES / USP
Pesquisadores da rede ONSA (Organização para Seqüenciamento e Análise de Nucleotídeos) identificaram 200 novos genes associados aos estágios de vida do verme Schistosoma mansoni, causador da esquistossomose, doença típica de países pobres, que atinge no mundo todo 200 milhões de pessoas e, só no Brasil, cerca de 10 milhões de habitantes. Com as conclusões que emergem da análise do genoma do parasita, estudado também por grupos de pesquisa em Minas Gerais,abrem-se novas perspectivas de combate à doença, hoje tratada com medicamentos que reduzem a quantidade de parasitas no sangue, mas não evitam a reinfecção – e há evidências de que os parasitas se tornam resistentes às drogas em uso.
Os achados sobre o Schistosoma ocorrem num momento particularmente fértil do Programa Genoma-FAPESP, inaugurado em 1997 com o mapeamento da bactéria Xylella fastidiosa, uma das pragas dos laranjais. No mês passado, foi concluído o genoma de outra bactéria, a Leifsonia xyli subsp xyli, que ataca a cana-de-açúcar (Saccharum officinarum) e reduz em até 27% a biomassa aproveitável para produção de açúcar e álcool. A Leifsonia é o primeiro projeto inteiramente nacional no âmbito de um programa filhote do Genoma-FAPESP, o Genomas Agronômicos e Ambientais (AEG), criado em 2000 a partir do seqüenciamento de uma variedade de Xylella que ataca as videiras, em conjunto com o Departamento de Agricultura dos Estados Unidos.
Chegam resultados importantes também do seqüenciamento de cloroplasto – uma estrutura celular – da cana-de-açúcar, que exibe uma impressionante semelhança com o milho (Zea mays), e está praticamente finalizado o de uma alga marinha, a Gracilaria tenuistipitata, produtora de ágar-ágar, um gel de larga aplicação industrial. O cloroplasto possui seu próprio genoma, cuja manipulação permitiria criar plantas transgênicas mais seguras. À medida que as informações se acumulam, é possível conhecer, num ritmo cada vez mais acelerado, a função de cada gene dos organismos estudados e os pontos mais vulneráveis dos causadores de doenças, seja em seres humanos, seja em plantas, a partir dos quais se estabelecem rotas de combate.
É esse o principal objetivo do trabalho com o Schistosoma, verme de 6 a 10 milímetros de comprimento e 0,5 milímetro de diâmetro que chega aos seres humanos por meio dos caramujos Biomphalaria glabrata. As equipes de São Paulo e de Minas que trabalham para deter seu avanço reforçam os projetos internacionais de identificação e análise dos genomas de agentes de doenças típicas de países subdesenvolvidos, como os protozoários Plasmodium falciparum, causador da malária, o Trypanosoma cruzi, responsável pela doença de Chagas, e a Leishmania, da leishmaniose.
Em pouco mais de um ano, um grupo da rede ONSA coordenado por Sergio Verjovski-Almeida, do Instituto de Química da USP – em colaboração com outros laboratórios da própria USP, da Universidade de Campinas (Unicamp), e dos institutos Butantan e Adolfo Lutz – produziu 130 mil seqüências das regiões expressas (atuantes) dos genes – as chamadas ESTs ou etiquetas de seqüências expressas, que são os fragmentos de DNA (ácido desoxirribonucléico) com as informações para a produção de proteínas. Essas seqüências de genes refletem os diversos estágios de vida do Schistosoma – adulto, ovo, miracídio, germball (a fase em que vive dentro do caramujo), cercária e esquistossômulo. Mas representam apenas uma parte da bagagem genética: enquanto o genoma da maioria dos parasitas tem de 10 a 30 milhões de pares de bases (unidades químicas do DNA), estima-se que o desse verme tenha 300 milhões de pares – e um número ainda indeterminado de genes.
Os pesquisadores paulistas identificaram seqüências completas de 200 novos genes, aos quais se atribui alguma função, por apresentarem similaridade com os de outros organismos, e outros 1.500 estão quase completos. Encontraram também cerca de 20 mil fragmentos totalmente novos, sem similaridade com os de outros organismos. Entre os genes de interesse, estão os ligados à produção de proteínas da superfície do verme. A equipe da USP obteve a seqüência completa de uma delas, que parece estar ligada ao escape do verme às defesas do organismo humano – pode, portanto, ser um caminho para a produção de uma vacina. Contra essa doença, cujo impacto social só perde para a malária, usa-se um medicamento à base de um fármaco chamado praziquantel. Aplicado em larga escala no Senegal, gerou cepas (variedades) de parasitas resistentes, caracterizadas em outubro de 2001 nos Annals of Tropical Medicine and Parasitology.
“A Organização Mundial da Saúde recomenda que se busque uma vacina, ainda que demore alguns anos”, diz Verjovski, “porque o remédio funciona somente depois que uma pessoa está infectada, mas não quebra o ciclo da doença.” Em dias ensolarados, o verme deixa os caramujos e ganha a água de lagos com águas paradas ou com pouca correnteza. Entra pela pele nos seres humanos, ganha a circulação sangüínea e se instala no fígado. Ali, transforma-se em adulto e se reproduz. Os ovos causam a destruição do fígado, que impede a circulação do sangue e faz o ventre inchar – é quando se caracteriza a barriga d’água, o nome popular da doença. Os ovos chegam aos intestinos e, eliminados por meio das fezes, caem na água na qual circulam os caramujos – e o ciclo recomeça.
Em Minas, um grupo coordenado por Guilherme Oliveira, do Centro de Pesquisa René Rachou, da Fundação Oswaldo Cruz (Fiocruz), concluiu em 2001 o seqüenciamento de 16.000 ESTs. Agora está em fase de implantação outro projeto, realizado pela Fiocruz e por universidades mineiras, com financiamento da Fundação de Amparo à Pesquisa do Estado de Minas Gerais (Fapemig) e do Conselho Nacional de Desenvolvimento Científico e Tecnológico (CNPq). Os pesquisadores almejam chegar a mais 50 mil seqüências.
Avançam também as análises comparativas da carga genética dos organismos. Dessas comparações surgem os indícios de processos evolutivos ocorridos ao longo de milhões de anos, que definem tanto as semelhanças quanto as diferenças entre as linhagens dos seres vivos. Os grupos paulistas descobriram que, curiosamente, o Schistosoma é geneticamente mais similar à mosca-de-frutas (Drosophila melanogaster) do que a outro verme cujo genoma já foi seqüenciado, o Caenorhabditis elegans.
Mais informações surpreendentes vêm à tona com as comparações estabelecidas a partir da Leifsonia xyli subsp.xyli, que causa perdas anuais da ordem de R$ 50 milhões em apenas uma das variedades de cana suscetíveis. “A Leifsonia é muito parecida com Streptomyces e Mycobacterium, dois gêneros que abrigam espécies patogênicas de animais”, comenta Luís Eduardo Aranha Camargo, pesquisador da Escola Superior de Agricultura Luiz de Queiroz (Esalq) e um dos coordenadores do seqüenciamento, co-financiado pela Cooperativa dos Produtores de Cana, Açúcar e Álcool do Estado de São Paulo (Copersucar).
A alta similaridade do genoma da Leifsonia – com 2.584.462 pares de bases e cerca de 2.600 genes, tamanho semelhante ao da Xylella – com a Streptomyces pode ter um valor comercial imediato: a Streptomyces é considerada uma biofábricade antibióticos, muitos dos quais também são encontrados na Leifsonia. Daí se explica o interesse em patentear genes muito semelhantes aos que produzem dois antibióticos, a estreptomicina e a nisina. Espera-se também chegar a novos paradigmas de interação planta-patógeno a partir da análise do genoma de Leifsonia. “Talvez essa bactéria inicie o processo de infestação na cana-de-açúcar usando um sistema secretor (pelo qual a bactéria patogênica exporta suas toxinas para fora da célula) semelhante ao da Mycobacterium, que causa tuberculose e lepra”, cogita Camargo.
As análises comparativas revelam sutis distinções genéticas entre bactérias da mesma espécie, mas com vítimas diferentes. É o caso das duas variedades de Xylella fastidiosa, a que ataca a laranjeira, num trabalho pioneiro encerrado em 2000, e a da videira – ou Xylella PD, assim chamada por causar a doença de Pierce, que ataca a região de vinhos nobres na Califórnia, nos Estados Unidos, e causa prejuízos estimados em US$ 40 milhões por ano. Concluído em meados do ano passado, o seqüenciamento da Xylella PD estabelece interessantes paralelos com a cepa da bactéria que infecta os laranjais do interior paulista, gerando a Clorose Variegada de Citrus (CVC), praga também chamada de amarelinho – responsável pela erradicação de 10 milhões de laranjeiras em 2001 e prejuízos estimados em R$ 650 milhões pelo Fundo Paulista de Defesa da Citricultura (Fundecitrus), co-financiador do seqüenciamento.
Os pesquisadores identificaram uma seqüência de 70 mil pares de bases encontrados só na Xylella dos citros. Parte dessa seqüência também está na Xanthomonas citri, causadora do cancro cítrico, mas não foi detectada nem naXylella da uva, nem na Xanthomonas campestris, que ataca repolhos. A cepa de Xylella que ataca a uva é, aliás, menor do que a causadora da praga do amarelinho: com cerca de 2,5 milhões de pares de bases, tem 200 mil pares de bases a menos. “Essa distinção pode ser conseqüência da história evolutiva da bactéria, o resultado da interação da bactéria na planta e desta com o meio ambiente”, deduz Marie-Anne Van Sluys, do Instituto de Biociências da USP e uma das coordenadoras do seqüenciamento da Xylella da uva.
Na área agrícola, abrem-se perspectivas de atuação com os projetos de seqüenciamento do cloroplasto. Essa estrutura, localizada no citoplasma das células vegetais, contém um minigenoma: também possui informação genética, como os cromossomos do núcleo celular. Pode ser possível criar plantas transgênicas alterando apenas o cloroplasto, e de uma forma mais segura para o meio ambiente, pois mais de 95% das espécies vegetais não têm cloroplastos nos grãos de pólen, nos quais se encontram as células sexuais masculinas. As modificações efetuadas nesse minigenoma, portanto, não seriam transmitidas para outras plantas via pólen.
Helaine Carrer, da Esalq, coordena um dos trabalhos pioneiros no uso desse método no Brasil: é o projeto do genoma do cloroplasto da cana-de-açúcar, com 141.182 pares de bases, no âmbito do projeto Genoma Cana. Helaine já pode dizer que o genoma do cloroplasto da cana tem maior similaridade com o genoma do cloroplasto do milho do que o de outras gramíneas, como o arroz (Oryza sativa) e o trigo (Triticum vulgare). “O conteúdo gênico observado no cloroplasto de cana é idêntico ao de milho, inclusive no arranjo posicional dos genes”, diz ela. Regiões intergênicas exclusivas do milho mostraram-se presentes em cana com similaridade maior que 98%. “É a maior similaridade entre cloroplastos já descrita até o momento”, enfatiza.
O segredo dessa incrível semelhança pode estar no mecanismo de fotossíntese desenvolvido ao longo do processo evolutivo da planta. Tanto a cana quanto o milho são plantas de fotossíntese C4 (isto é, o primeiro produto formado no processo de fotossíntese tem quatro carbonos), enquanto arroz e trigo são plantas C3. “Enquanto os genomas nucleares da cana e do milho exibem muitas diferenças devido aos processos evolutivos de cada espécie, os genomas cloroplastidiais mostraram-se praticamente idênticos”, afirma Helaine. A descoberta tem uma aplicação: qualquer diferença no cloroplasto pode servir de marcador nas pesquisas de melhoramento genético.
Mariana Cabral de Oliveira, do Instituto de Biociências da USP, faz um trabalho semelhante com a Gracilaria tenuistipitata, alga marinha de origem asiática que produz ágar-ágar, gel utilizado nas indústrias alimentícia, farmacêutica e cosmética. A importância econômica desse organismo foi um dos motivos que o elegeram como objeto de estudo, além do fato de essa espécie ser de cultivo fácil em laboratório e freqüentemente empregada para estudos em fisiologia de organismos fotossintetizantes. “É quase um organismo modelo”, diz Mariana. Iniciado em outubro de 2000, o seqüenciamento do cloroplasto da alga, com 183.883 pares de bases, revelou um grupo de genes relacionados à fotossíntese, cuja manipulação ampliaria a produtividade da alga.
Mariana dedica-se também ao seqüenciamento de ESTs da Gracilaria, em colaboração com pesquisadores da USP e da Suécia. Já foram seqüenciadas 3.000 ESTs – uma pequena parte do genoma do organismo, que possui de 24 a 32 cromossomos, de acordo com a espécie. Não se sabe a que tamanho pode chegar o genoma nuclear dessa alga, mas já se conhecem espécies de algas com genomas maiores do que o humano, que tem 3 bilhões de pares de bases. A garimpagem de genes promete outras surpresas.
A cana bem compreendida
O genoma das variedades de cana-de-açúcar mais cultivadas no Brasil nunca foi analisado sob tantas abordagens quanto nos 37 artigos científicos que compõem a mais recente edição da revista brasileira Genetics and Molecular Biology, da Sociedade Brasileira de Genética, dedicada aos resultados do projeto Genoma Cana ou Sucest, financiado pela FAPESP e Copersucar.Os artigos – produzidos por cerca de 200 pesquisadores de 39 equipes da rede ONSA – discutem variados aspectos do projeto e dos mais de 43 mil clusters, regiões do genoma da cana que podem ter um ou mais genes, identificados pelo Sucest. “Esses trabalhos trazem informações importantes não só para a compreensão da biologia da cana, mas também de outras gramíneas cultivadas de alto valor comercial, como o milho, arroz e sorgo”, diz Paulo Arruda, da Universidade Estadual de Campinas (Unicamp), coordenador do projeto.
Alguns trabalhos abrem perspectivas rumo a uma possível manipulação genética com o intuito de melhorar o rendimento do cultivo de cana, aumentar a resistência a doenças ou reduzir os custos de produção. A equipe de Adriana Hemerly, da Universidade Federal do Rio de Janeiro (UFRJ) identificou um grupo de genes que parece estar associado à relação simbiótica (benéfica) entre duas bactérias fixadoras de nitrogênio, Gluconacetobacter diazotrophicus e Herbaspirillum rubrisubalbicans, e a cana. Quando se instalam no interior da planta, elas fixam o nitrogênio, o que naturalmente reduz a necessidade do uso de adubo nitrogenado. “Queremos aprimorar essa relação de simbiose”, diz ela.
Os primeiros resultados são animadores. Após inocular as duas espécies de bactérias na cana, Adriana e seus colegas (da Embrapa Agrobiologia, do Rio, e também da Unicamp) mediram a expressão (o nível de uso) dos genes da planta em várias situações. Viram que 274 genes da base de dados do Sucest se expressavam apenas em plantas com aG. diazotrophicus, 198 eram usados somente quando a cana abrigava a H. rubrisubalbicans e 62 se mostravam ativos só quando esse cultivo apresentava simultaneamente as duas bactérias. “Antes do Sucest conhecíamos quatro ou cinco genes relacionados à simbiose.” Numa outra linha de trabalho, uma equipe coordenada por Marcelo Menossi, da Unicamp, identificou 43 genes que podem estar relacionados à tolerância da planta ao alumínio, cujo excesso reduz a produtividade. “Esses genes talvez possibilitem o controle da resistência ao alumínio na planta”, diz Menossi.
Outro artigo da Genetics de relevância para a biotecnologia trata da identificação de SNPs, um tipo de mutação, no genoma da cana, uma planta altamente complexa em cujo DNA pode haver até 10 cópias de um mesmo gene. SNPs é a sigla em inglês de single nucleotide polymorphism, ou polimorfismo de um único nucleotídeo, a unidade química que forma a seqüência genética. O termo designa, portanto, mutações em trechos de genes que são causadas pela troca de apenas um nucleotídeo. A maioria dessas mutações tende a ser inofensiva, mas outras podem afetar a aparência ou alterar o comportamento da planta. Se bem conhecidos e mapeados, os SNPs podem ser usados como marcadores moleculares.
Num trabalho com resultados ainda preliminares, pesquisadores da Unicamp deram uma pequena prova de que os dados do Sucest são úteis para a montagem de um eventual mapa de SNPs da cana: analisaram dois genes que codificam a enzima álcool desidrogenase, já conhecidos em outras espécies, nos quais identificaram 40 SNPs. “É muito difícil diferenciar os alelos (cópias) de um gene da cana”, diz Laurent Grivet, pesquisador do Cirad, instituto francês especializado em agricultura tropical, que trabalha hoje na Unicamp. “Acho que, na cana, não vai ser possível relacionar os SNPs ao seu fenótipo (aparência da planta), mas devemos usar esses polimorfirmos como marcadores moleculares.”
Os projetos
1. Genoma Xylella fastidiosa PD; Modalidade Subprograma Genomas Agronômicos e Ambientais; Coordenadoras Marie-Anne Van Sluys e Mariana Cabral de Oliveira – USP; Investimento US$ 925.181,84
2. Genoma Leifsonia xyli; Modalidade Subprograma Genomas Agronômicos e Ambientais; Coordenador Luís Eduardo Aranha Camargo e Cláudia Barros Monteiro Vitorello – USP; Investimento US$ 1.150.841,14
3. Genoma Schistosoma mansoni; Modalidade Programa Genoma – FAPESP; Coordenador Sérgio Verjovski-Almeida – USP; Investimento US$ 1.244.697,50
4. Genoma Gracilaria tenuistipitata; Modalidade Linha regular de auxílio à pesquisa; Coordenadora Mariana Cabral de Oliveira – USP; Investimento R$ 68.000,00 e US$ 34.000,00
5. Genoma Cloroplasto da Cana-de-Açúcar; Modalidade SUCEST – Projeto de Seqüenciamento de EST de Cana-de-Açúcar; Coordenador; Helaine Carrer – USP; Investimento R$ 49.954,45 e US$ 207.341,15

