
CRISTIANO MASCARO
Plantação no interior paulista: a citricultura é uma das bases da economia do estado mais rico do paísCRISTIANO MASCAROA Xylella fastidiosa é a essa altura uma velha conhecida dos biólogos e dos brasileiros que acompanham as narrativas da produção científica nacional, independentemente de seu campo de atuação. Mas a bem pesquisada doença que essa bactéria produz em laranjeiras e outras plantas cítricas no Brasil, a clorose variegada dos citros (CVC), mais conhecida por praga do amarelinho, ainda resiste às investidas dos cientistas que insistem há mais de uma década em decifrá-la integralmente. “Sabemos como começa a doença e como a bactéria ocupa os vasos da planta e neles se expande, porém não somos capazes de controlar eficazmente o estabelecimento da doença”, diz Marie-Anne Van Sluys, professora titular no Departamento de Botânica da Universidade de São Paulo (USP) e chefe do Laboratório de Genômica e Elementos de Transposição (GaTE-Lab) da mesma universidade. Entretanto, como o método científico não se deixa paralisar pelo desconhecimento e faculta a quem o segue a proposição simultânea de diferentes hipóteses para um mesmo problema – ou o cerco a um mesmo objeto por distintas abordagens –, há hoje, em paralelo, “uma forte percepção de que o momento-chave para o controle da doença é quando a bactéria começa a produzir o biofilme”, prossegue Marie-Anne.
Esse biofilme, pouco ressaltado quando a X. fastidiosa viveu seu grande momento de estrela, no centro do projeto pioneiro das pesquisas genômicas no Brasil, em 2000, é um material de aparência gelatinosa que se deposita junto à parede do vaso da planta, encostado a uma outra substância que ela também produz: a goma xantana. Esta, sim, por seu visível papel no entupimento dos vasos e consequente impedimento da livre passagem da seiva no xilema da planta, foi destacada como elemento importante para a caracterização da CVC no artigo científico que tornou público o genoma completo da X. fastidiosa. “A sensação que temos hoje é de que, se for rompida a produção do biofilme no momento em que a bactéria começa a produzi-lo ou se essa produção for afetada de algum modo, teremos uma boa condição para o controle da doença”, comenta Marie-Anne, hoje coordenadora adjunta da FAPESP na área de Ciên-cias da Vida.
O controle stricto sensu, diga-se, era um alvo desejável, espécie de cereja do bolo, mas não estava entre os objetivos centrais do projeto de sequenciamento do genoma da Xylella fastidiosa, apresentado com justiça em seu lançamento como um empreendimento destinado a revolucionar a ciência brasileira. Naquela manhã de 14 de outubro de 1997, o público de professores, pesquisadores e empresários que superlotava o auditório da FAPESP soube com detalhes que a ambiciosa iniciativa na qual a Fundação investiria US$ 12 milhões, valor espantoso para o financiamento de um projeto de pesquisa na época e para o qual o Fundo de Desenvolvimento da Citricultura (Fundecitrus) aportaria mais US$650 mil, tinha como primeira meta sequenciar o genoma completo do patógeno e descrever os novos achados científicos obtidos nesse processo. Tratava-se, portanto, de fazer ciência na fronteira do conhecimento, levando a bom termo um projeto de biologia molecular.

REPRODUÇÃOCapa da Nature de 13 de julho de 2000: reconhecimento internacional ao empreendimento brasileiroREPRODUÇÃO
O segundo objetivo era formar pesquisadores altamente qualificados, em grande escala e em curto intervalo de tempo, ampliando em muito a competência da pesquisa paulista e brasileira em biologia molecular. E o terceiro era mobilizar a comunidade científica para estudar problemas socioeconômicos significativos que ela poderia ajudar a resolver, como a preocupante praga do amarelinho, àquela altura. Mal se poderia supor ali, entre as falas que se sucediam no quarto andar da sede da Fundação, a boa dose de ousadia, autoconfiança e persistência que seria requerida nos próximos meses das pessoas e instituições que iriam se embrenhar pelo indevassado terreno da genômica no Brasil. “As chances de dar errado eram enormes”, lembra sorrindo o hoje empresário e então diretor científico da FAPESP, o físico José Fernando Perez, um dos principais artífices do belo voo da biologia molecular no país que o projeto da X. fastidiosa representa. “Aposto que foi o projeto de sequenciamento de genoma que começou pior preparado no mundo”, disse depois de tudo concluído, com muito humor, o coordenador de DNA do projeto, o inglês Andrew Simpson.
Passados 28 meses dessa solenidade de lançamento, em 21 de fevereiro de 2000 os 192 pesquisadores que tinham recém-concluído com sucesso e explosões de alegria o sequenciamento do genoma da X. fastidiosa viram-se aplaudidos com estonteante entusiasmo por mais de mil pessoas reunidas numa festa na bela Sala São Paulo, enquanto recebiam troféus, medalhas e diplomas do mérito científico e tecnológico instituídos especialmente para a ocasião pelo governo do estado de São Paulo (é bem verdade, registre-se, que a mais prolongada ovação da noite foi endereçada a Perez, espécie de regente titular da obra).
Já em 13 de julho de 2000, dois anos e nove meses após o lançamento do projeto que ao longo desse período levantara algumas críticas acerbas de quem achava que sequenciar genoma não era fazer ciência, em meio aos mais numerosos comentários sobre o salto científico que ele poderia representar para o país, a Nature destinou sua capa ao projeto da Xylella. A revista publicou na edição 6.792, volume 406, além do artigo científico de sete páginas com os achados originais da pesquisa, assinado por 116 cientistas, uma notícia detalhada a respeito do estudo na seção News and Views e um editorial no qual sintetizou o projeto pioneiro da genômica brasileira como “uma realização política tanto quanto científica”.
É uma definição precisa. Primeiro, o projeto da X. fastidiosa inequivocamente gerou conhecimento científico que continua se desdobrando, 12 anos depois de concluído o sequenciamento da bactéria. E, em paralelo, ele cumpriu seu objetivo anunciado de ampliar rapidamente a capacitação brasileira em biologia molecular, ao mesmo tempo que serviu de âncora a vários outros projetos em genômica que vieram a compor o Programa Genoma FAPESP. Mais: o projeto ajudou a ressituar o Brasil no mapa da produção científica internacional e contribuiu decisivamente para um redesenho da imagem do país na mídia interna e externa quanto à sua competência para criar conhecimento científico. Não bastassem esses dividendos políticos, a arquitetura do projeto, o ritmo em que foi tocado e o uso de um novo meio pelo qual se fariam as trocas de informações e dados entre pesquisadores de três dezenas de laboratórios (a internet) tiveram um impacto evidente no modo de se produzir ciência em São Paulo desde então, trazendo-o para a velocidade contemporânea. E, por fim, o projeto foi o ponto de partida para a criação de empresas relacionadas ao negócio da genômica, spin-offs hoje absorvidas pelo mercado.
Um bom roteiro para resgatar a partir deste ponto alguns traços essenciais da experiência da Xylella pode começar pelas contribuições à genômica contidas no artigo publicado pela Nature e ressaltadas na edição 55 de Pesquisa FAPESP, de julho de 2000. Nele está registrado que ali se trata do 24º genoma completo de bactéria conhecido pela ciência e do primeiro de um fitopatógeno. O sequenciamento mostrou que o micro-organismo investigado possui 2.679.305 pares de bases nitrogenadas ou nucleotídeos em seu cromossomo.
 O cromossomo carrega 2.904 genes, regiões codificadoras de proteínas, dos quais um terço era novo para a ciência em 2000. Do total, 47% dos genes tiveram suas funções precisamente descritas pelo grupo brasileiro, percentual que se situou um pouco abaixo daqueles que outros grupos de pesquisa de genoma de bactéria haviam obtido – por exemplo, 54% dos genes com funções descritas na Thermotoga marítima, 52,5% na Deinococcus radiodurans e 53,7% na Neisseria meningiditis. Os autores atribuíram o resultado mais baixo à inexistência prévia de qualquer outra sequência completa de genoma de bactéria fitopatogênica.
O cromossomo carrega 2.904 genes, regiões codificadoras de proteínas, dos quais um terço era novo para a ciência em 2000. Do total, 47% dos genes tiveram suas funções precisamente descritas pelo grupo brasileiro, percentual que se situou um pouco abaixo daqueles que outros grupos de pesquisa de genoma de bactéria haviam obtido – por exemplo, 54% dos genes com funções descritas na Thermotoga marítima, 52,5% na Deinococcus radiodurans e 53,7% na Neisseria meningiditis. Os autores atribuíram o resultado mais baixo à inexistência prévia de qualquer outra sequência completa de genoma de bactéria fitopatogênica.
Na virada do século, quando o Brasil era produtor de quase metade do suco de laranja concentrado distribuído no mercado internacional, a receita nacional da citricultura alcançava US$2 bilhões anuais; as receitas de exportações giravam em torno de US$ 1,6 bilhão por ano e a atividade gerava no estado de São Paulo 400 mil empregos diretos e indiretos. Nesse cenário, o adoecimento das laranjeiras por CVC, que já atingia 34% dos laranjais paulistas, produzia prejuízos estimados em US$ 100 milhões anuais. Os dados de 2009 mostram uma receita total da atividade ampliada para US$ 6,9 bilhões anuais, exportações em torno de US$ 3,15 bilhões, um número bem menor de empregos diretos e indiretos, ou seja, 230 mil postos de trabalho e a CVC atingindo proporção idêntica dos laranjais paulistas, isto é, 35% deles.
No artigo da Nature abordava-se o metabolismo refinado X. fastidiosa, com sua adaptação para o uso de açúcares encontrados livres na seiva do xilema e da glicose derivada da quebra da celulose das paredes das células vegetais. Descoberta de peso do projeto foi a identificação dos genes que codificam moléculas envolvidas na adesão da célula. Tais moléculas, anteriormente só vistas em patógenos de humanos e de outros animais, encontram-se na superfície celular da bactéria, respondem pela aderência ao tecido epitelial nos hospedeiros, e encontrá-las na X. fastidiosa ampliou a evidência de que os mecanismos de patogenicidade das bactérias são os mesmos, infectem elas plantas, animais ou seres humanos.
Os desdobramentos atuais das hipóteses e observações daquele momento provaram que elas estavam corretas, e novas descobertas foram feitas. O grupo de pesquisa liderado por Marcos Machado e Alessandra de Souza, da Agência Paulista de Tecnologia Agrícola – Instituto Agronômico de Campinas (Apta-IAC) identificou a existência de células persistentes nos vasos, o que resulta em parte na dificuldade de controle e persistência da doença no campo. Eles também demonstraram a ocorrência de duas formas de vida da bactéria: a fase planctônica e a fase de biofilme. A eles se juntam os trabalhos realizados pelo grupo de Aline Silva, do Instituto de Química (IQ) da USP, que demonstram a importância da adesão da bactéria aos vasos e da sinalização por ferro, reforçando a observação da existência de 67 genes dedicados à retirada de ferro e outros metais da seiva da planta.
A síntese do que fizeram os pesquisadores brasileiros está clara nas conclusões finais do artigo da Nature. Eles determinaram “não só o metabolismo básico e as características de replicação da bactéria, mas também numerosos mecanismos potenciais de patogenicidade. Alguns jamais tiveram sua ocorrência antes postulada para fitopatógenos, fornececendo novas percepções para a generalidade desses processos”. Os resultados obtidos permitirão, diz o artigo, o começo de uma detalhada comparação entre patógenos animais e vegetais. E, para completar, “as novas informações deverão fornecer bases para uma investigação experimental, acelerada e racional das interações entre a X. fastidiosa e seus hospedeiros, que devem conduzir a novos achados nas abordagens para o controle da CVC, a famosa praga do amarelinho”.
Essa terminologia do paper estava ainda no lugar do sonho quando ao abrir a solenidade do dia 14 de outubro de 1997, o então presidente da FAPESP e hoje seu diretor científico, o físico Carlos Henrique de Brito Cruz, classificou o genoma da Xylella como “um projeto singular, destinado a marcar um lugar na história da ciência e da tecnologia no estado de São Paulo e no Brasil”. Justificou essa visão antecipatória valendo-se de argumentos que integram um pensamento que vem refinando ao longo dos anos sobre a construção das sociedades do conhecimento. Assim, observou que o projeto iria reunir aspectos da ciência básica e estudos na fronteira do conhecimento teórico com trabalhos de pesquisa aplicada e desenvolvimento tecnológico. “Com esse projeto vai se fazer o que tem sido uma das grandes preocupações da FAPESP e de outras instituições de ciência e tecnologia em São Paulo e no Brasil: aliar a ciência à produção, aproximar a ciência do PIB, da riqueza nacional, do desenvolvimento social e econômico”, disse. Seguiu por essa via até qualificar o projeto como “revolucionário”.

FABIO MELO FONTES/FUNDECITRUSA cigarrinha transmissora do amarelinhoFABIO MELO FONTES/FUNDECITRUS
As origens e a arquitetura do empreendimento da X. fastidiosa foram apresentadas ao público pelo diretor científico, José Fernando Perez, não sem ele antes recorrer a alguns números para dar suporte à sua visão de que se tornara urgente e imprescindível a montagem de projeto com tal dimensão. Divulgados havia pouco tempo pelo Ministério da Ciência, Tecnologia e Inovação, tais números indicavam que, enquanto a participação brasileira em geral na produção científica mundial, registrada na base de dados do Institute of Scientific Informations (ISI), praticamente dobrara entre 1981 e 1995, passando de 0,44% para 0,82%, o desempenho em biologia molecular mostrara um avanço muito lento. Em outros termos, se o número total de artigos brasileiros indexados crescera a um fator de 2,12, ante 1,35 para a produção científica mundial, em biologia molecular a relação entre os dois fatores estava longe de confortável: 1,69 para a produção brasileira e 1,89 para a produção mundial. Observe-se já aqui, a propósito, que se em 1996 os artigos de biologia molecular correspondiam a 4,20% do total de artigos científicos brasileiros registrados na base de dados Scopus, em 2007 eles representavam 6,68%, com sua produção apresentando crescimento contínuo a partir de 2002.
Os números exibidos por Perez, articulados ao argumento de que barraria o desenvolvimento desejável para o país deixar que se ampliasse a defasagem brasileira numa área científica essencial a várias outras, ditas pura ou aplicada, pareciam destinados a refrear de saída as críticas que grupos da comunidade científica ensaiavam contra o novo investimento da FAPESP. Não refrearam. Por um bom período a Fundação teve que entrar em debates exibindo outros números para provar que não havia concentração perversa de seus investimentos na pesquisa genômica, em detrimento dos demais campos científicos.
Uma década mais tarde, em palestra num ciclo organizado por Pesquisa FAPESP paralelamente à exposição “Revolução Genômica”, montada pelo Instituto Sangari no atual Pavilhão das Culturas Brasileiras, no Parque do Ibirapuera (ver Pesquisa FAPESP, suplemento especial de setembro de 2008), Perez observou que de 1997 a 2003, a FAPESP investiu, no conjunto dos 20 projetos que integraram seu Programa Genoma, US$ 39 milhões. “O investimento nunca passou de 2,4% do orçamento da Fundação”, enfatizou. Por outro lado, as contrapartidas aportadas por instituições e empresas parceiras das diferentes iniciativas do programa atingiram US$11,7 milhões. Isso incluiu os aportes do Fundecitros, na Xylella, da Coopersucar, no genoma da cana (ver reportagem na página 54), do Departamento de Agricultura dos Estados Unidos, na Xylella da uva, do Instituto Ludwig, no genoma do câncer, além das empresas Suzano, Ripasa, Voto-rantim e Duraflora, no projeto genoma do eucalipto, Embrapa, no genoma do café, e Central Bela Vista, no projeto genoma do boi.
Há quem veja por outro ângulo as críticas dirigidas ao projeto da X. fastidiosa, caso de seu primeiro mentor, ao lado de Perez, e depois um de seus coordenadores, o biólogo Fernando Reinach. “O projeto era revolucionário”, disse em longa entrevista pingue-pongue na edição 100 da Pesquisa FAPESP. “Encontrou uma resistência muito grande do pessoal mais velho (…). Dizia-se que não era ciência. Era, enfim, a resistência à mudança. E só conseguimos fazer o projeto porque os jovens que tinham conseguido entrar no sistema já não estavam sob o domínio do pessoal mais velho. Quando lançamos o edital para a seleção dos laboratórios, foi esse pessoal novo que disse ‘eu faço’. Quando os mais velhos disseram ‘nós não vamos entrar’, isso foi muito sintomático do que estava ocorrendo.”

FABIO MELO FONTES/FUNDECITRUSA laranja menor foi colhida de uma laranjeira infectada pela XylellaFABIO MELO FONTES/FUNDECITRUS
Detalhes saborosos sobre a gênese do projeto da X. fastidiosa, ilustrativos de como se dá no mundo real o processo de decisões que impulsionam a produção do conhecimento ou as mudanças no ambiente da produção científica, apareceram en passant na solenidade de lançamento. Só mais adiante, contados por Perez ou Reinach, eles se tornariam elementos da história do aparentemente mais narrado projeto de pesquisa brasileira, dentro e fora do país.
“Precisávamos de uma ideia nova que proporcionasse uma mudança na biotecnologia brasileira, criando competência”, relembrou Perez na palestra de 2008. Ele a percebia como uma área estratégica com capacidade para responder “às características econômicas, à biodiversidade, à agricultura, à pecuária e a problemas de saúde pública específicos do país”. Eram nesse sentido suas conversas com os assessores no começo de 1997, em especial com Fernando Reinach, então professor titular de bioquímica na Universidade de São Paulo (USP) e um dos coordenadores de área da diretoria científica da Fundação. E foi como continuação dessas conversas que ocorreu um telefonema decisivo de Reinach para Perez.
“Num final de semana, em 1º de maio de 1997, eu estava no sítio em Piracaia e pensei: em vez de fazer um projeto de infraestrutura, vamos fazer um projeto de genoma, juntar todo mundo num objetivo único. Era uma ideia, para mim, muito estranha. Eu liguei para o Perez, que estava em Santos, e ele veio até o sítio. Conversamos e a ideia se cristalizou.”
Já na semana mesmo após o feriado, Perez iniciou as gestões para organizar o projeto. O micro-organismo a ser sequenciado ainda não estava escolhido, mas om certeza seria uma bactéria, com um genoma grande o suficiente para permitir o envolvimento de muita gente no trabalho e pequeno o bastante para não inviabilizá-lo. Dos Estados Unidos, onde então trabalhava três meses por ano, Reinach enviou pouco tempo depois a Perez, a seu pedido, o primeiro rascunho do projeto. Segundo o ex-diretor científico da FAPESP, todas as ideias fundamentais que seriam usadas na estrutura que sustentaria o projeto já estavam lá.
As discussões sobre o projeto foram incorporando novos protagonistas, ao mesmo tempo que o assunto era levado a debate no conselho superior da FAPESP. Paulo Arruda, biólogo, professor da Universidade Estadual de Campinas (Unicamp) e Marcos Machado, do Centro de Citricultura Sylvio Moreira, estavam entre os primeiros participantes das discussões. Mais adiante Ricardo Brentani sugeriu a Perez que incorporasse ao grupo o pesquisador Andrew Simpson, inglês que estava há alguns anos no Brasil. Era preciso ter consultores internacionais de peso já na fase da discussão das características do projeto e Paulo Arruda levou à FAPESP Goffeau, entre outras qualificações, muito experiente em bioinformática. Aliás, essa era uma área-chave num projeto genoma e poderia se tornar crítica, dada a experiência praticamente nula do país em suas técnicas. Uma das ideias propostas por Goffeau foi que se fizesse essa parte do trabalho na França. Mas Reinach sugeriu uma conversa do grupo com dois jovens do Instituto de Computação da Unicamp, João Setúbal e João Meidanis, que já vinham lidando com bioinformática em simulação de genomas e tinham publicado um livro sobre o assunto, ainda que nunca houvessem trabalhado com genoma real. “Por isso havia uma insegurança no ar. Os assessores internacionais não acreditavam que conseguiríamos resolver os problemas da bioinformática, mas foi ela um dos grandes sucessos do projeto”, comentou Perez numa de suas avaliações posteriores do empreendimento da Xylella.
O grupo desses assessores externos, o steering committee, era composto, além de Goffeau, por Steve Oliver e John Sgouros, também britânico. E foi numa reunião com Oliver, na FAPESP, que se definiu a estrutura de comando do projeto: haveria dois laboratórios centrais a que se remeteriam todos os laboratórios envolvidos no trabalho de sequenciamento, mais o laboratório de bioinformática e um coordenador geral. O edital de chamada aos laboratórios, publicado pouco depois do lançamento do projeto, seguiu exatamente essa orientação ao definir as vagas. Foi assim que Andrew Simpson tornou-se o coordenador geral de DNA do projeto, com a responsabilidade inclusive de garantir clones da bactéria para todos os laboratórios, Reinach e Arruda foram escolhidos coordenadores de sequenciamento e Meidanis e Setúbal, coordenadores de bioinformática.
A seleção dos 30 laboratórios, de quase uma centena que atendeu à chamada do edital, considerou, segundo Reinach, que 10 deles precisavam ter algum conhecimento prévio de sequenciamento, outros 10 deveriam ter pesquisadores ligados à agricultura, com alguma noção sobre a Xylella, e mais 10 poderiam ser comandados por pesquisadores que nem conheciam a bactéria nem tinham prática de sequenciamento, “mas eram pessoas inquestionavelmente competentes em termos científicos, sempre com vontade de aprender mais, perfil, por exemplo, de José Eduardo Krieger”.
Na entrevista que concedeu à Pesquisa FAPESP depois de concluído o sequenciamento (Especial “O futuro da genômica no Brasil”, edição 51, março de 2000), Simpson, que tinha orgulho de seu papel no projeto e já comemorara muito o fato de o Brasil ter “entrado numa festa seleta sem ter sido convidado e ter feito bonito”, detalhou como foi difícil superar o desconhecimento geral em relação à X. fastidiosa e lembrou, sempre com fino humor, outros obstáculos que tiveram de ser superados, uma vez começado o trabalho. “Havia um coordenador que nunca sequenciara um genoma, não trabalhara com bactéria e sequer sabia o que era a Xylella fastidiosa. Havia um coordenador de informática que nunca havia lidado com um projeto de DNA na vida e uma equipe espalhada por todo o estado de São Paulo. Vários não sabiam quase nada de biologia molecular. Pior: ninguém no estado tinha a bactéria viva, muito menos DNA, muito menos ainda a biblioteca de DNA. Começamos sem qualquer evidência de que o trabalho iria funcionar”. Segundo Simpson, foi a promessa de Bové de fornecer a bactéria e o DNA necessário que deu confiança para começar. “No final, Marcos Machado, do Instituto Agronômico de Campinas (IAC), deu todo o DNA que eu logo usei no projeto.”
Mesmo admitindo a falta de condições prévias para fazer o que tinha sido anunciado, Simpson não considerava uma “loucura de brasileiro” esse começo, onde até a ligação por internet para desenvolver um projeto que dependia crucialmente dela apresentava ainda uma enorme precariedade. “Foi confiança na habilidade da comunidade de executar qualquer tarefa dada. Não há nada de loucura.” Para Simpson, o momento mais difícil entre todos foi a fase final, em que era preciso vencer os gaps que restavam na sequência montada. “Primeiro, eu não tinha a menor ideia de como fazer. Nunca havia feito e os papers não contam exatamente como fazer. Era preciso inventar uma solução. Segundo, tínhamos muito pouca informação sobre a Xylella antes de começar o sequenciamento. Não sabíamos sequer o tamanho dos gaps, porque não sabíamos o tamanho total do genoma”. Por isso, como na época observou Marie-Anne, encaixadas em 6 de janeiro de 2000 as últimas peças do gigantesco quebra-cabeça, uma palavra, espécie de senha repetida ao telefone, marcou sobre todas a comemoração que se repetiu por 35 laboratórios espalhados pelo território paulista: “Fechou!”.
O salto imenso que deixava definitivamente no passado os dias de manejo desajeitado e lento dos sequenciadores importados no começo de 1998 se completara quando o pesquisador Luís Eduardo Aranha Camargo enviou o read (parte da biblioteca de clones) que fechou o genoma. E tornou-se público quando Simpson anunciou esse fechamento aos cientistas reunidos no I Encontro de Genomas Microbianos Relevantes para a Agricultura, promovido pelo Departamento de Agricultura dos Estados Unidos, em San Diego, Califórnia, nos dias 8 e 9 de janeiro. O Brasil ganhara uma expertise até então dominada apenas por 14 outros grupos de pesquisa nos Estados Unidos, Europa e Japão.
Hoje, uma busca simples pela X. fastidiosa para o período 1995/maio de 2012 na base de dados PubMed, referência em publicações de qualidade na área de medicina, faz retornar 343 artigos que a citam, dos quais 330 publicados depois do paper dos brasileiros na Nature. Deles, 110 artigos, ou seja, um terço do total, têm como endereço o Brasil. Trata-se de contribuição notável para o conhecimento do tema.
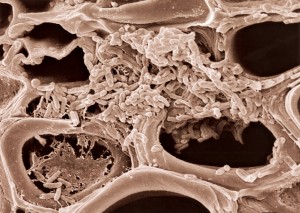
ELLOT W. KITAJIMA/USP
Colônia de bactérias Xylella infesta e entope a parede de vasos que transportam a seiva da laranjeiraELLOT W. KITAJIMA/USP“As predições e hipóteses elaboradas naquele nosso primeiro artigo foram sendo demonstradas ao longo dos anos”, comenta Marie-Anne Van Sluys. Hoje se indaga sobre a origem da CVC e o papel do biofilme na doença. “Há razões fortes para suspeitarmos de que o controle pode se dar pela intervenção na formação do biofilme – retoma o raciocínio Marie-Anne –, há uma patente depositada pelo grupo coordenado por Marcos Machado e Alessandra Alves (INPI: 018110011623 PCT: BR/2012/000003 Data depósito: INPI 31/03/2011 PCT: 9/1/2012) que faz uso de um análogo de cisteína para bloquear esse processo, estratégia já utilizada com sucesso para patógenos de humanos. Por outro lado, dada a enorme quantidade de vírus que são encontrados nos genomas das várias linhagens de Xylella, resultados observados a partir dos estudos de expressão por microarray realizados pelos grupos da doutora Suely Gomes (IQ-USP) e doutora Marilis do Vale Marques (ICB-USP), em conjunto com o sequenciamento de múltiplas linhagens de Xylella coordenado pela doutora Aline Silva, sugerem que condições ambientais possam induzir a multiplicação das partículas virais até matar a bactéria. “Se o vírus sair do genoma e se propagar formando capsídeos virais, ele pode explodir a célula, matando a bactéria”, ela imagina.
No ambiente bem fecundado pelo projeto pioneiro da genômica, grupos de pesquisa que ali se consolidaram formaram novas lideranças e estabeleceram sólidas colaborações internacionais, indagam-se hoje sobre isso e muito mais: o grupo original de Marcos Machado, no IAC, agora com Alessandra Alves de Souza na coordenação da linha de pesquisa com a famosa bactéria, por exemplo, debruça-se sobre a interação com a planta. O grupo de Marilis do Valle Marques tem trabalhado na obtenção de mutantes da X. fastidiosa, úteis para Aline Maria da Silva, do Departamento de Bioquímica da USP, e lidera o estudo genômico de diferentes cepas da X. fastidiosa na América do Sul, três das quais infestam citros e as demais, cafeeiros, ameixeira e hibiscos, respectivamente, avançando na pesquisa de genes patogênicos da bactéria.
Além da continuidade da pesquisa, vale citar como outros atestados do sucesso do empreendimento da Xylella, a criação, por pesquisadores ligados ao projeto, das empresas Alellyx, que levou tanto os recursos dos genomas como outras ferramentas da biotecnologia para a agricultura, Scylla, que presta serviços em bioinformática, e CanaVialis, especializada em novas variedades de cana-de-açúcar. A primeira e a terceira foram compradas a seu principal investidor, o fundo Votorantim Novos Negócios, também formado na esteira do projeto pioneiro da genômica e dirigido por Reinach, e em 2008 pela Monsanto.
O editorial da Nature sobre a X. fastidiosa em 2000 observou, entre outros pontos, que o sucesso do projeto da X. fastidiosa, somado ao fato incomum de uma agência do mundo avançado e industrializado – o Departamento de Agricultura dos Estados Unidos – ter contratado a pesquisa genômica de uma variante da Xylella de um país em desenvolvimento, “endossa a determinação do Brasil de entrar na idade pós-genômica de mãos dadas com os cientistas dos países mais ricos”.
Os Projetos
1. Projeto Genoma – Fapesp: Laboratório de Sequenciamento (nº 1997/13451-6) (1997-2000); Modalidade Programa Genoma; Coordenador Andrew John George Simpson – Instituto Ludwig; Investimento R$ 1.329.975,72
2. Projeto Genoma – Fapesp: Laboratório de Sequenciamento (nº 1997/13457-4) (1997-2000); Modalidade Programa Genoma; Coordenador Fernando de Castro Reinach – USP; Investimento R$ 1.535.926,46
3. Projeto Genoma – Fapesp: Laboratório de Sequenciamento (nº 1997/13475-2) (1997-2000); Modalidade Programa Genoma; Coordenador Paulo Arruda – Unicamp; Investimento R$ 932.244,71
4. Projeto Genoma – Fapesp: Laboratório de Sequenciamento (nº 1997/13463-4) (1997-2000); Modalidade Programa Genoma; Coordenador Jesus Aparecido Ferro – Unesp; Investimento R$ 1.534.700,66
Artigo científico
SIMPSON, A.J. et al. The genome sequence of the plant pathogen Xylella fastidiosa. The Xylella fastidiosa Consortium of the Organization for Nucleotide Sequencing and Analysis. Nature. v. 406, n. 6792, p.151-59, 2000.
SILVA, A.C. R. da et al. Comparison of the genomes of two Xanthomonas pathogens with differing host specificities. Nature. v. 417, n. 6887, p. 459-63, 2002.
De nosso arquivo
O salto quântico da ciência brasileira – Edição nº 100 – junho de 2004
Da Xylella à Alellyx – Edição nº 74 – abril de 2002
As descobertas se multiplicam – Edição nº 60 – dezembro de 2000
Bravo, cientistas! – Edição nº 51 – março de 2000
Xylella – concluído o genoma da bactéria – Edição nº 50 – janeiro e fevereiro de 2000
Brasil se afirma no seleto clube da genômica mundial – Edição nº 48 – novembro de 1999
Os primeiros resultados – Edição nº 29 – março de 1998
Um projeto para revolucionar a ciência brasileira – Edição nº 25 – outubro de 1997
