Uma notícia promissora no combate às bactérias resistentes a medicamentos surgiu em maio de 2023. Com o auxílio de inteligência artificial, pesquisadores do Canadá e dos Estados Unidos identificaram um composto com ação potente e específica contra Acinetobacter baumannii, uma bactéria que frequentemente causa infecções graves nos pulmões e no trato urinário de pessoas internadas em hospitais. Esse patógeno integra a lista criada em 2017 pela Organização Mundial da Saúde (OMS) das bactérias contra as quais se buscam novos tratamentos com urgência porque os antibióticos atuais já não conseguem eliminá-las.
A equipe liderada por James Collins, do Instituto de Tecnologia de Massachusetts (MIT), nos Estados Unidos, e Jonathan Stokes, da Universidade McMaster, no Canadá, treinou um modelo computacional para reconhecer as propriedades de 480 compostos com alguma ação contra A. baumannii entre os 7.684 já testados para combater o microrganismo. Depois, apresentou ao modelo outras 6.680 moléculas sem ação antibiótica conhecida, a fim de que o programa encontrasse aquelas com maior probabilidade de matar o patógeno. Em poucas horas, a lista de 6.680 compostos foi reduzida para 240, algo que levaria dias ou meses pelos métodos tradicionais de testagem. Quarenta das 240 moléculas foram capazes de inibir o crescimento de A. baumannii em laboratório. Uma em especial saiu-se bem – a abaucina – e seguiu para os testes em animais. O caminho ainda é longo até que possa vir a se tornar um antibiótico para uso humano.
Ainda que reduzam o tempo gasto na triagem de compostos, a modelagem computacional e a inteligência artificial, por si só, não devem resolver a questão da resistência aos antibióticos. O problema é tão antigo quanto o uso desses compostos pela medicina moderna e, na avaliação de especialistas, exigirá ações diversas: do emprego rigorosamente controlado desses medicamentos na saúde humana e animal à prevenção de infecções por meio de higiene e, quando possível, de vacinação, além de, claro, o desenvolvimento de novos medicamentos.
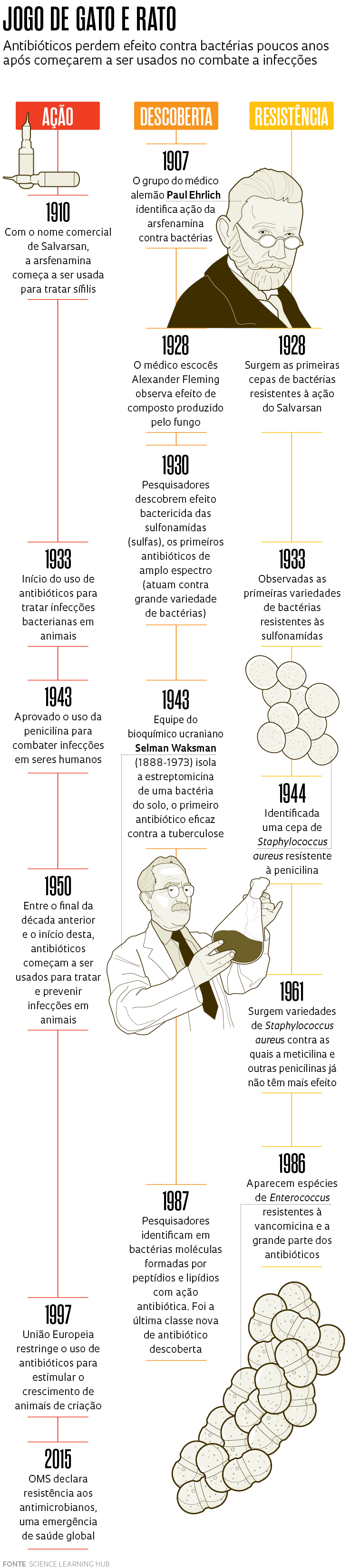
O primeiro antibiótico sintético de que se tem notícia foi a arsfenamina, um composto contendo arsênico identificado em 1907 no laboratório do médico alemão Paul Ehrlich (1854-1915), embora haja pistas de que preparos com produtos naturais fossem usados desde a Antiguidade.
Em testes com coelhos em 1909, Ehrlich e o bacteriologista japonês Sahachiro Hata (1873-1938) demonstraram que a arsfenamina era capaz de eliminar a bactéria da sífilis (Treponema pallidum) sem matar os animais. O trabalho abriu caminho para que, no ano seguinte, o composto, com o nome comercial de Salvarsan, passasse a ser usado no tratamento dessa doença sexualmente transmissível que acompanhava a humanidade havia séculos. Em 1928, porém, surgiram os primeiros relatos de casos em que o Salvarsan não surtia mais efeito contra algumas variedades de T. pallidum.
O mesmo ocorreu com a penicilina. O composto foi identificado em 1928 pelo médico escocês Alexander Fleming (1881-1955) em uma cultura da bactéria Staphylococcus aureus contaminada por acaso com um fungo do gênero Penicillium em seu laboratório no Saint Mary’s Hospital, em Londres, e logo passou a ser usado para tratar infecções. Ainda levaria mais de uma década para que outros pesquisadores conseguissem purificar o princípio ativo e dominar a produção em grande escala. No entanto, mesmo antes do uso amplo da penicilina na Segunda Guerra Mundial, quando salvou a vida de milhares de soldados, já havia sinais de que as bactérias poderiam se tornar resistentes ao medicamento.
Fleming sabia disso e passou a chamar a atenção para o problema, inclusive no discurso feito ao receber o Nobel de Medicina de 1945. “Não é difícil tornar micróbios resistentes à penicilina em laboratório, expondo-os a concentrações insuficientes para matá-los… Existe o perigo de que o homem ignorante possa facilmente subdosar-se e, ao expor os seus micróbios a quantidades não letais da droga, torná-los resistentes”, afirmou o médico escocês.
Tem sido assim. O uso disseminado desses compostos na saúde humana, animal e na produção agrícola vem sendo acompanhado do surgimento de bactérias resistentes a diferentes antimicrobianos. “As previsões de Fleming revelaram-se precisas: a utilização incorreta, por vezes um verdadeiro abuso, de antibióticos, acelera o desenvolvimento e a propagação de bactérias resistentes a eles”, escreveram o químico Marco Terreni, da Universidade de Pávia, na Itália, e seus colaboradores em um artigo de revisão sobre novos antibióticos, publicado em 2021 na revista Molecules.

USDA-ARSAndrew Moyer (1899-1959), que descobriu como produzir penicilina em grande escalaUSDA-ARS
“É como um jogo de xadrez”, afirma a farmacêutica bioquímica Ilana Camargo, coordenadora do Laboratório de Epidemiologia e Microbiologia Moleculares (LEMiMo), instalado no campus de São Carlos da Universidade de São Paulo (USP). Seu grupo se dedica a caracterizar bactérias resistentes a múltiplos antibióticos e a identificar novos compostos com o potencial de combatê-las. O mais recente encontrado por eles é um derivado sintético da plantaricina 149, peptídeo produzido pela bactéria Lactobacillus plantarum. Nos testes in vitro, a molécula eliminou 60 bactérias de diferentes espécies e linhagens com graus variáveis de resistência a medicamentos já disponíveis no mercado, segundo artigo publicado em fevereiro de 2023 na Antibiotics. “Sempre que pensamos ter colocado uma bactéria ‘em xeque’ com um novo antibiótico, logo ela encontra uma forma de escapar”, conta.
Desde o início do século XX, dezenas de antibióticos já foram identificados. São moléculas que pertencem a diferentes classes e apresentam mecanismos de ação distintos. Uma proporção importante delas – superior a 75%, segundo alguns pesquisadores – é de origem natural, produzida por outros microrganismos. Por um longo período, uma das campeãs em fornecer novos antibióticos foram as bactérias do gênero Streptomyces. Um artigo de revisão publicado em 2019 por pesquisadores do Reino Unido na revista Current Opinion in Microbiology estima que essas bactérias, encontradas no solo e em vegetação em decomposição, foram a fonte de 55% dos antibióticos descobertos entre 1945 e 1978. Neomicina, estreptomicina, grisemicina e cloranfenicol estão entre eles. Quase sempre, no entanto, pouco tempo após a introdução de um novo composto no mercado, as bactérias se tornam resistentes a ele. Em alguns casos, a mais de um antibiótico (ver linha do tempo).
Até o final dos anos 1970, as grandes empresas da indústria farmacêutica solucionavam a questão lançando um novo antibiótico, produzido com base em moléculas extraídas de outras bactérias ou fungos e com estrutura química diferente dos anteriores. Mas esses compostos começaram a escassear porque se tornou mais difícil encontrá-los nos microrganismos mais estudados. “Os frutos ao alcance da mão já haviam sido colhidos”, conta Camargo, que integra o Centro de Pesquisa e Inovação em Biodiversidade e Fármacos (CIBFar), um dos Centros de Pesquisa, Inovação e Difusão (Cepid) apoiados pela FAPESP. “Passou a ser necessário investir em pesquisas para modificar a estrutura química de moléculas já conhecidas”, explica.
A facilidade anterior de obter novos antibióticos e o bom desempenho dos então disponíveis criavam uma falsa sensação de segurança, que se somou a uma mudança importante na indústria farmacêutica. No início dos anos 1980 uma nova geração de gestores chegou à direção das grandes empresas e reorientou o foco dos investimentos para o desenvolvimento de medicamentos mais caros e lucrativos, destinados a tratar o câncer e doenças associadas ao estilo de vida, como o diabetes, relataram o químico Matthew Todd, da University College London, no Reino Unido, e colaboradores em 2021 na revista Wellcome Open Research. “O problema central do pipeline de antibióticos vazio não é científico, mas econômico”, afirmaram.
O retorno advindo da venda dos novos fármacos, mais caros e difíceis de se obter, em geral, não pagava o custo do desenvolvimento. E, segundo alguns especialistas, os lucros com as vendas caíam ainda mais com a chegada ao mercado das versões genéricas desses medicamentos. “Nesse período, várias grandes indústrias fecharam departamentos inteiros e encerraram programas de triagem e desenvolvimento de antibióticos”, lembra a bioquímica brasileira Andréa Dessen, coordenadora do Grupo de Patogênese Bacteriana do Instituto de Biologia Estrutural (IBS), em Grenoble, França.