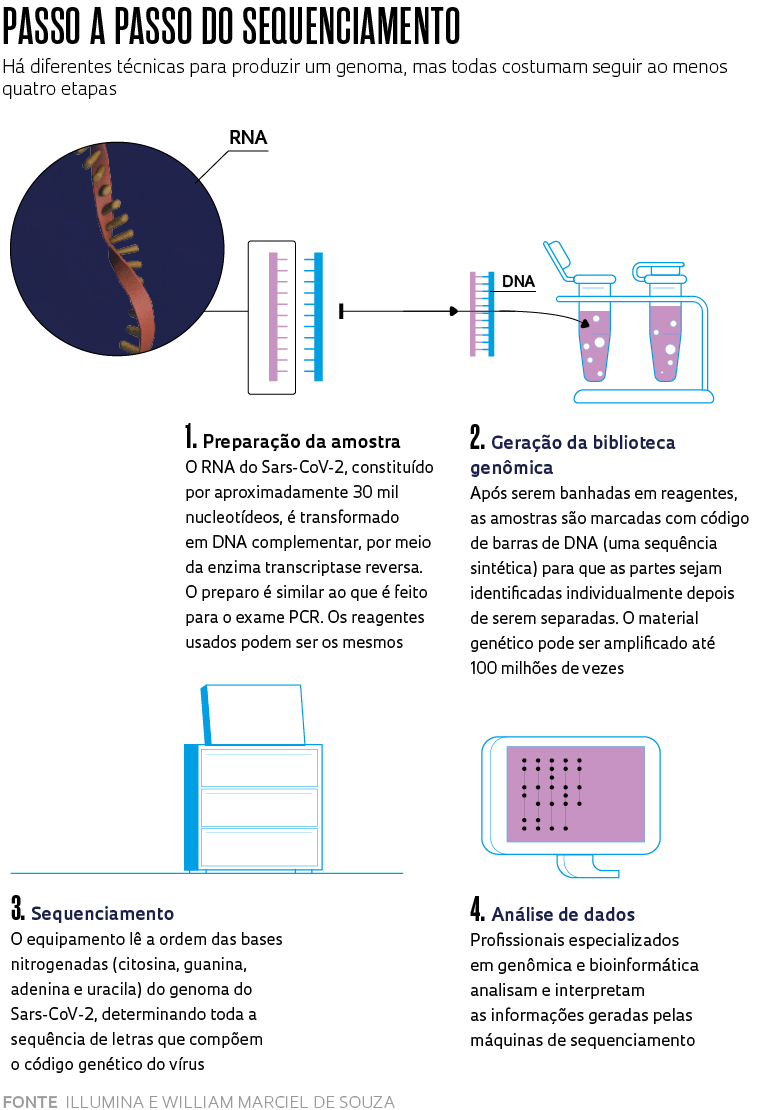
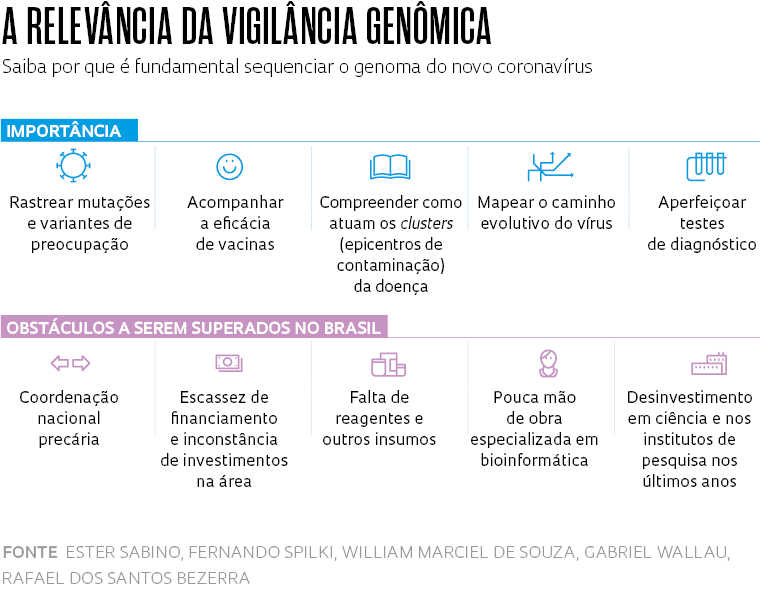
Conhecer o genoma do vírus presente na população infectada com o Sars-Cov-2 é essencial para rastrear mutações e mapear as cepas que circulam no país. Também é importante para entender como atuam os clusters (epicentros de contaminação) de Covid-19, a doença causada por ele, assegurar a acurácia de testes diagnósticos e a eficácia de vacinas e reconstruir a filogenia (o caminho evolutivo) do patógeno. O Brasil, contudo, segue no vermelho em termos de vigilância genômica do Sars-CoV-2 15 meses após a confirmação do primeiro caso da doença. Apesar dos esforços de pesquisadores que trabalham na área, apenas 0,059% dos quase 15 milhões de casos positivos em território brasileiro para o novo coronavírus registrados até 20 de maio teve seu genoma sequenciado e depositado na plataforma Gisaid, principal banco internacional de compartilhamento de dados sobre o código genético e as mutações detectadas no patógeno.
“As 5 mil amostras sequenciadas são muito pouco para um ano inteiro de epidemia”, afirma a médica Ester Sabino, coordenadora do Centro Conjunto Brasil-Reino Unido para Descoberta, Diagnóstico, Genômica e Epidemiologia de Arbovírus (Cadde), financiado pela FAPESP e pela agência britânica Medical Research Council. A declaração de Sabino, professora do Instituto de Medicina Tropical (IMT) da Faculdade de Medicina da Universidade de São Paulo (FM-USP), foi dada durante audiência pública da Comissão Externa de Enfrentamento à Covid-19 da Câmara dos Deputados, realizada em abril.
Naquela ocasião, os dados do Gisaid mostravam que o Brasil havia depositado 5.488 do 1.050.224 de genomas completos compartilhados na plataforma desde janeiro de 2020 – 0,52% dos sequenciamentos realizados no planeta. O número aumentou para 9.316 na segunda quinzena de maio, mas a proporção em relação ao total mundial pouco se alterou, o que representa uma parcela ínfima, considerando-se que o país respondia por 9,6% dos casos confirmados de Covid-19 do mundo e por 12,6% dos óbitos.
Em números absolutos, 1,58 milhão de genomas do novo coronavírus já havia sido sequenciado por 172 países até 20 de maio. Os Estados Unidos apareciam em primeiro lugar, com 451 mil genomas compartilhados, superando o Reino Unido (408 mil), que durante muito tempo manteve a liderança. Em termos proporcionais às infecções registradas de Covid-19 no país, o ranking era encabeçado por Islândia, que sequenciou 77,7% dos casos positivos, Austrália (59,1%) e Nova Zelândia (46,4%).
A importância de se fazer a vigilância genômica ganhou maior destaque no país depois da identificação, em janeiro, da variante de preocupação P1, em Manaus. A nova cepa espalhou-se rapidamente pela cidade, provavelmente a partir de novembro de 2020, mesmo com estimados 67% da população tendo sido expostos ao Sars-CoV-2 na primeira onda da pandemia, em abril e maio de 2020. Os cientistas calculam que a P1 seja entre 1,7 e 2,4 vezes mais transmissível do que as registradas até então na região e pode escapar à imunidade formada por infecção prévia.
Em cerca de sete semanas, a fração das amostras do novo coronavírus classificada como P1 no município saiu de zero para 87%, informaram Sabino e colegas em artigo publicado na revista Science em 14 de abril deste ano. O estudo também confirmou, como suspeitavam os pesquisadores, que o segundo surto em Manaus está associado ao surgimento e à rápida disseminação da nova variante de preocupação (VOC). Essa é a designação dada pela Organização Mundial da Saúde (OMS) e autoridades sanitárias nacionais às cepas que trazem maior risco potencial à saúde pública, seja porque são mais transmissíveis, virulentas ou alteram a sintomatologia da doença.
A P1 integra o grupo de variantes que mais preocupam governos e cientistas do mundo nos primeiros meses de 2021. As outras são a B.1.1.7, detectada pela primeira vez na Grã-Bretanha, a B.1.351, identificada na África do Sul, e a B.1.617, que contribuiu para que os casos da doença explodissem na Índia (ver Pesquisa FAPESP nº 302) e também foi classificada como de preocupação. Em sua fala na Câmara dos Deputados, Sabino destacou que, sem a expansão da vigilância genômica no Brasil, não será possível fazer frente às novas variantes que certamente ainda irão surgir. Os principais entraves para isso, argumenta a pesquisadora, são a ausência de uma coordenação nacional de resposta à epidemia e a falta de recursos para sequenciamento.
No país, duas redes nacionais interligam instituições e pesquisadores no esforço de decodificar o genoma do Sars-CoV-2, identificando as linhagens presentes nos diversos estados e mutações genéticas. A Rede Genômica Fiocruz é apoiada pelo Ministério da Saúde e coordenada por cientistas da Fundação Oswaldo Cruz (Fiocruz). Já a Rede Nacional de Ômicas da Covid-19 (Rede Corona-ômica BR) recebe recursos do Ministério da Ciência, Tecnologia e Inovações (MCTI) e reúne pesquisadores de instituições como USP, Universidade Estadual de Campinas (Unicamp), Laboratório Nacional de Computação Científica (LNCC), no Rio de Janeiro, entre outras.
No estado de São Paulo, foi estabelecida em março a Rede de Alerta de Pandemia de Variantes Emergentes do Sars-CoV-2, coordenada pelo Instituto Butantan, com a participação de equipes da USP, Universidade Estadual Paulista (Unesp), Faculdade de Medicina de São José do Rio Preto (Famerp) e da empresa privada Mendelics, especializada em análises genéticas. Em uma amostra colhida em março na cidade de Sorocaba, pesquisadores da rede detectaram pela primeira vez no país a presença da variante sul-africana B.1.351. “Confirmamos duas vezes: pelas mutações e pela árvore filogenética”, disse o biomédico e bioinformata Rafael dos Santos Bezerra, do Hemocentro de Ribeirão Preto, um dos autores de artigo, ainda sem a revisão de pares, que descreve a detecção da linhagem por monitoramento genômico.
Participantes dessas redes projetam para os próximos meses uma escalada no número de sequenciamentos e na capacidade de o país realizar a vigilância genômica. “Os laboratórios da Rede Corona-ômica receberam lotes maciços de insumos no fim de março. Esperamos um grande incremento no sequenciamento nas próximas semanas”, informa o virologista Fernando Spilki, coordenador da rede e professor da Universidade Feevale, em Nova Hamburgo (RS). De acordo com ele, o material é suficiente para produzir cerca de mil genomas – quase um quinto do que foi depositado no primeiro ano da pandemia.
Material, no caso, são os kits com os reagentes necessários para preparar as amostras e fazer “correr” as máquinas de sequenciamento. Esses insumos são, em sua maioria, importados dos Estados Unidos e da Europa, assim como os sequenciadores, cujo número de unidades aumentou no país a partir da eclosão da epidemia de zika vírus em 2015. Poucas empresas fabricam os equipamentos e os respectivos kits. Uma das maiores é a norte-americana Illumina, cujos aparelhos são conhecidos pela confiabilidade dos resultados. Outra é a britânica Oxford Nanopore Technologies, que comercializa o sequenciador portátil MinION.
“Todos os grupos estão enfrentando dificuldades de recebimento dos reagentes para poder dar uma resposta em escala”, confirma o biólogo Gabriel Wallau, do Instituto Aggeu Magalhães, da Fiocruz, em Recife. “Como os Estados Unidos passaram a sequenciar bastante e o mundo inteiro também está fazendo o mesmo, precisamos de uma boa argumentação com a empresa para conseguir os reagentes.”
Após assumir a presidência norte-americana, no início do ano, Joe Biden definiu a vigilância genômica como prioridade. Nos primeiros meses de 2021, o número de sequenciamentos mais do que dobrou. Ainda assim, em maio a porcentagem de sequenciamento atingia apenas 1,5% dos casos positivos para Covid-19 naquele país – abaixo dos 5% preconizados em artigo preprint publicado na plataforma medRxiv como o ideal para detectar variantes emergentes antes que elas superem 1% dos casos de determinado lugar.
“Possuímos know-how como qualquer outro país, além de termos equipamentos de qualidade, mas há dois problemas principais interligados: faltam recursos para o sequenciamento e, quando eles existem, há um descompasso para a obtenção dos reagentes, que nem sempre chegam em tempo hábil”, avalia Wallau. O laboratório em que ele atua em Pernambuco estava finalizando a compra de reagentes no começo de maio. “Acredito que em breve teremos uma visão bem melhor da evolução do novo coronavírus no país.”
O município de São Paulo passará a contar em breve com um programa de monitoramento de variantes de preocupação dentro de um projeto desenvolvido no âmbito do Cadde, o projeto binacional coordenado por Sabino. A ideia da iniciativa, articulada pela FAPESP em entendimento com a prefeitura, é analisar semanalmente cerca de 380 amostras positivas para o patógeno, escolhidas aleatoriamente, de forma proporcional à população de cada região da cidade. Em parte das amostras, será feito o sequenciamento completo do genoma do vírus; em outra, um tipo de exame PCR que consegue identificar a presença das linhagens mais preocupantes conhecidas. No programa-piloto, realizado na primeira semana de março, 71,2% das 73 amostras testadas de moradores do município eram de variantes de preocupação. Dessas, 64,4% eram P1, a variante de Manaus, e 6,8% a B.1.1.7, encontrada inicialmente no Reino Unido.
No Brasil, muitos laboratórios que fazem o sequenciamento genômico do novo coronavírus também realizam o diagnóstico da doença por PCR ou têm convênio com instituições públicas e particulares que o fazem, como os Laboratórios Centrais de Saúde Pública (Lacen). Esses acordos facilitam o acesso às amostras a serem sequenciadas.
O sequenciamento completo do genoma é o padrão ouro para a descoberta de mutações e de novas variantes de interesse e preocupação do Sars-CoV-2, mas uma nova ferramenta entrou no radar dos pesquisadores brasileiros: a genotipagem baseada em PCR por tempo real, exame usado na detecção do vírus. “Um dos protocolos hoje utilizados foi criado pela equipe do imunologista Felipe Naveca, da Fiocruz Amazonas”, informa Spilki. Pelo protocolo desenvolvido, o teste molecular consegue dizer se uma amostra positiva para o novo coronavírus pertence a uma das linhagens de preocupação já conhecidas. “Assim, conseguimos avaliar não apenas centenas, mas milhares de sequências de vírus, praticamente tudo o que já está circulando. E obtemos resultados sobre a dispersão das variantes de uma maneira mais rápida.”
Tecnologia similar foi desenvolvida no Laboratório de Biologia Integrativa do Instituto de Ciências Biológicas da Universidade Federal de Minas Gerais (ICB-UFMG). O baixo preço e a rapidez dos resultados são vantagens desse novo método para detecção de variantes, de acordo com a equipe liderada pelo geneticista Renato Santana. O teste custa em média R$ 70 e fica pronto em quatro horas, enquanto um sequenciamento pode custar a partir de R$ 500 e levar semanas para ser concluído.
Outro gargalo importante para expandir o trabalho de vigilância genômica no país, segundo Wallau, é a escassez de especialistas em bioinformática. “Uma coisa é sequenciar; outra é analisar a sequência. As análises mais refinadas demandam tempo”, afirma o pesquisador. “Temos máquinas muito poderosas que geram um enorme volume de dados, mas como analisar todos esses dados de maneira precisa?”
Paulo Chapchap, presidente do Conselho de Administração do Instituto Todos pela Saúde (ITpS), concorda. “Temos no país alguns centros importantes de bioinformática dedicados a doenças infecciosas, mas precisamos formar mais profissionais”, diz. Apoiado pela Fundação Itaú Unibanco, o ITpS dispõe de R$ 200 milhões para investir em iniciativas voltadas à vigilância genômica.
Os cortes de investimento em ciência, tanto na pesquisa básica quanto na aplicada, nos últimos anos no Brasil, também afetam as investigações. “Houve um forte desinvestimento, prejudicando universidades e centros de pesquisa, não só do governo federal”, diz o biomédico William Marciel de Souza, que faz estágio de pós-doutorado na Universidade de Oxford, no Reino Unido, e tem colaborado com grupos brasileiros nos trabalhos de análise dos genomas sequenciados no país. “O que está sendo feito agora em termos de sequenciamento pela Fiocruz, Rede Vírus e alguns pesquisadores pelo país é um milagre. É um esforço fundamental para se tentar entender a pandemia no país e ajudar a controlá-la.”
Projetos
1. Centro Conjunto Brasil-Reino Unido para Descoberta, Diagnóstico, Genômica e Epidemiologia de Arbovírus (Cadde) (nº 18/14389-0); Modalidade Projeto Temático; Pesquisadora responsável Ester Cerdeira Sabino (USP); Investimento R$ 5.271.740,75.
2. Avaliação do impacto de vírus emergentes e re-emergentes em hemoterapia e transplante de células-tronco por meio de técnicas moleculares avançadas (nº 17/23205-8); Modalidade Jovem Pesquisador; Pesquisador responsável Svetoslav Nanev Slavov (USP); Investimento R$ 1.534.184,25.
Artigos científicos
FARIA, N. et al. Genomics and epidemiology of the P.1 Sars-CoV-2 lineage in Manaus, Brazil. Science. 14 abr. 2021.
SLAVOV, S. N. et al. Genomic monitoring unveil the early detection of the Sars-CoV-2 B.1.351 lineage (20H/501Y.V2) in Brazil. medRxiv. 4 abr. 2021.
VAVREK, D. et al. Genomic surveillance at scale is required to detect newly emerging strains at an early timepoint. medRxiv. 15 jan. 2021.
GRUBAUGH, N. D. et al. PCR assay to enhance global surveillance for Sars-CoV-2 variants of concern. medRxiv. 12 mar. 2021.
Republicar