Vírus para combater infecções humanas, bactérias contra câncer, DNA programado como um código de computador. Soluções buscadas pela ciência podem ter sido desenvolvidas pela evolução em organismos unicelulares. Uma delas é a existência de vírus capazes de identificar e destruir bactérias específicas. São os bacteriófagos, ou fagos, cada vez mais reconhecidos como possível alternativa a antibióticos e fonte de aplicações biotecnológicas inovadoras. Outra frente de pesquisa, a biologia sintética, manipula o material genético dos microrganismos e direciona bactérias e leveduras a produzirem novos tratamentos, funcionar como sensores e abrir outras grandes – embora microscópicas – possibilidades.
Muitos desses projetos saem de pesquisa básica que busca compreender organismos e mecanismos sem inicialmente mirar aplicações ou produtos. É o caso do estudo microbiológico de composteiras do Parque Zoológico de São Paulo (ver Pesquisa FAPESP nº 181), cujo objetivo era catalogar a diversidade bacteriana e investigar processos de decomposição. Mas “onde há muitas bactérias, há muitos fagos”, diz a bioquímica Aline Maria da Silva, do Instituto de Química da Universidade de São Paulo (IQ-USP), que trabalha no projeto em parceria com o bioinformata João Carlos Setubal, do mesmo instituto. “Os fagos modulam as populações de bactérias”, explica ela, que no início se interessou pelos vírus unicamente por interferirem em seu objeto de estudo. Mas seu estudante de mestrado Deyvid Amgarten, um apaixonado por fagos, foi além e começou a analisar o material genético viral das composteiras que recebem alimentos e dejetos dos animais do zoológico.
v
Como uma ideia leva à outra e atiça a curiosidade, o grupo decidiu procurar por fagos úteis no combate a bactérias patogênicas, que provocam doenças. Havia na compostagem bactérias do gênero Pseudomonas, parentes da perigosa P. aeruginosa, causadora de infecções hospitalares. Em relatório publicado no início do ano, a Organização Mundial da Saúde classificou essa bactéria como a segunda em termos de urgência de pesquisa para novos antibióticos, diante da alta resistência às medicações existentes. A vantagem dos fagos é que evoluem em conjunto com seus alvos, mantendo a capacidade específica de atacar um tipo de bactéria sem prejudicar o organismo humano.
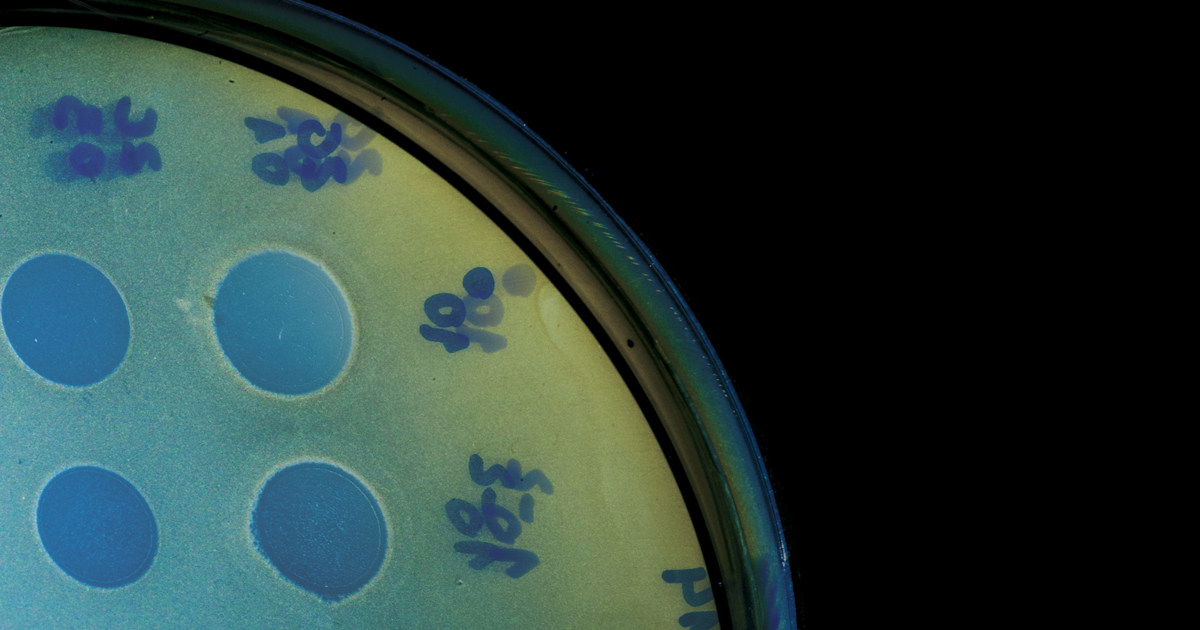
O grupo do IQ conseguiu isolar – e caracterizar geneticamente – três fagos distintos, conforme descreve em artigo publicado em maio na BMC Genomics. “Os fagos degradam o biofilme de bactérias em 24 horas”, afirma Aline. Para saber como esses vírus se comportam no ambiente e dentro do organismo de mamíferos, seria necessário que outros grupos de pesquisa se interessassem por fazer esses testes. Outros fagos já foram testados contra P. aeruginosa em camundongos com sucesso, conforme mostra estudo liderado pelo imunologista Aras Kadioglu, da Universidade de Liverpool, no Reino Unido, publicado na edição de julho da revista Thorax. O tratamento eliminou bactérias dos pulmões de 70% dos camundongos com uma infecção estabelecida – condição que em seres humanos é uma das principais causas de mortalidade hospitalar.

VICTOR BALCÃO / UNISO
Fagos mais claros já injetaram DNA em fragmento de bactériaVICTOR BALCÃO / UNISOO método para isolar os fagos no IQ-USP é o mesmo usado em aplicações médicas no leste da Europa. Desde 1923, o Instituto Eliava, em Tbilisi, Geórgia, usa agentes virais contra qualquer infecção bacteriana, inclusive aquelas que nenhum outro tratamento conseguiu eliminar (ver infográfico). Uma revisão publicada em 2001 na Antimicrobial Agents and Chemotherapy, por pesquisadores do Eliava e da Universidade de Maryland, nos Estados Unidos, compara o sucesso de tratamento por antibióticos (64%) e fagos (82%), e afirma que o tratamento viral é ainda mais eficaz (95%) quando aplicado por via intravenosa. Por isso, são procurados por pacientes do mundo todo. A história conta que a descoberta da possibilidade de usar vírus como tratamento foi concomitante à de antibióticos. Como a ação destes é mais abrangente, e por isso mais afeita à produção industrial, a estratégia ganhou precedência mundial – exceto no antigo bloco soviético, onde o isolamento imposto pela cortina de ferro exigia soluções criativas e independentes do Ocidente.
Ao longo das décadas de uso de antibióticos, a evolução tratou de selecionar bactérias resistentes e deu origem a linhagens responsáveis por graves casos de infecções hospitalares. No final de abril noticiou-se o caso do psiquiatra norte-americano Tom Patterson, da Faculdade de Medicina da Universidade da Califórnia em San Diego, que no início de 2016 entrou em coma por causa de uma infecção que não respondia aos antibióticos. Sua mulher, Steffanie Strathdee, e o médico Robert Schooley, ambos professores na mesma faculdade, apelaram para um último recurso e conseguiram autorização para tentar a terapia por fagos. “Amostras do paciente foram enviadas a dois grupos de pesquisa que selecionaram os fagos”, conta Schooley, que defende que os fagos sejam amplamente reconhecidos como parte do arsenal contra infecções bacterianas. “Empresas de vários países estão começando a trabalhar no desenvolvimento desse tipo de terapia”, afirma.
Patterson saiu do coma e seu caso, em análise para publicação em periódico científico, foi apresentado no último dia 27 de abril no Instituto Pasteur em Paris, durante o evento comemorativo do centenário da pesquisa com bacteriófagos. A data comemora a descoberta dos fagos pelo microbiologista franco-canadense Felix d’Herelle (1873-1949), do Pasteur, na mesma época em que o britânico Frederick Twort (1877-1950), e por isso foi batizada como Dia da Terapia com Fagos Humanos. D’Herelle, aliás, participou da fundação do Instituto Eliava, mas não voltou a Tbilisi depois que o bacteriologista George Eliava (1892-1937) foi executado pelo regime stalinista.
Enquanto na maior parte dos países a possibilidade de tratamento é restrita a casos considerados perdidos, pesquisadores se limitam a caracterizar esses abundantes e ubíquos vírus. A microbiologista Sylvia Cardoso Leão, da Universidade Federal de São Paulo (Unifesp) e especialista em micobactérias – as causadoras da tuberculose e de infecções difíceis de tratar –, recentemente se interessou pelos fagos. Em parceria com a microbiologista Cristina Viana Niero, do campus de Diadema da Unifesp e participante do projeto do zoológico, Sylvia e seu então estudante de mestrado James Daltro Lima-Junior buscaram nas composteiras fagos especializados nas micobactérias. Encontraram, mas não as bactérias com as quais interagem. Em colaboração com Aline e Setubal, o grupo caracterizou esses vírus do ponto de vista genético, que o biólogo molecular Graham Hatfull, da Universidade de Pittsburgh, nos Estados Unidos, comparou às sequências do acervo que mantém. “É a maior coleção do mundo de bacteriófagos que infectam micobactérias, uma ferramenta importante para entendermos melhor essas espécies”, afirma Sylvia. A surpresa foi verificar que um dos fagos paulistanos estudados é muito parecido com outro isolado de uma amostra coletada na África do Sul. Talvez uma contribuição de algum animal africano residente no zoológico.
Sylvia monitora infecções por micobactérias e já ajudou a estabelecer protocolos para combater surtos e detectar a causa do problema, como resistência a desinfetantes usados em hospitais. Ela defende que os fagos seriam um bom aliado nessa constante batalha. “Eles são o que há de mais comum, uma arma da natureza.” Hoje é possível comprar produtos à base de fagos para eliminar bactérias em verduras e reduzir o teor de Salmonella em galinhas, por exemplo, mas não para combater doenças humanas.
A capacidade dos fagos de reconhecer suas bactérias correspondentes também pode dar origem a sensores, como o que o engenheiro alimentar português Victor Balcão, da Universidade de Sorocaba (Uniso), no interior paulista, pretende fazer em parceria com a bioquímica Marta Vila, da mesma instituição. “A ideia é criar uma fita adesiva que se possa pregar na parede dos hospitais para detectar Pseudomonas aeruginosa”, explica. O projeto, batizado como PneumoPhageColor, envolve revestir esses adesivos com um gel recheado de fagos capturados na água do esgoto de hospitais. Se existirem bactérias no ar, a ideia é que elas penetrem no hidrogel e sua interação com o vírus e subsequente rompimento (um processo conhecido como lise) produza um brilho, como fazem vaga-lumes e alguns cogumelos (ver Pesquisa FAPESP nº 255), por meio de uma reação química entre substâncias integradas no hidrogel. O alerta pode indicar a necessidade de desinfetar um ambiente. “A probabilidade de se entrar em um pronto-socorro e sair com uma infecção é de 30%”, avisa o pesquisador.
São passos que ele espera percorrer até o fim do próximo ano, agora que montou o laboratório, e que têm como base conquistas obtidas em projeto anterior. “Conseguimos estabilizar o fago no gel por meio de aprisionamento seguido de uma reação química”, conta, conforme publicou em 2013 na Enzyme and Microbial Technology. Em seguida, o grupo de Sorocaba demonstrou que as bactérias levam 6 minutos para penetrar 3 milímetros no hidrogel, suficiente para entrarem em contato com os fagos, como mostrou em 2014 na Applied Biochemistry and Biotechnology.

SOONHEE MOON / UNIVERSIDADE COLUMBIA
Culturas de bactérias tingidas para estudo de morfologia se transformam em arte no laboratório de Tal DaninoSOONHEE MOON / UNIVERSIDADE COLUMBIAGenética sob controle
O horizonte de aplicações biotecnológicas com fagos é amplo e pode ir além de tirar proveito de suas habilidades naturais. Esses vírus podem ser geneticamente alterados para atuar como sensores de patógenos ou armas contra eles, ou para transportar sequências genéticas desejadas para dentro de células. Outra propriedade da interação entre vírus e bactérias é o sistema CRISPR-Cas9, algo como um sistema imunológico das bactérias que mantém um registro dos vírus com que já tiveram contato. O sistema deu origem a uma ferramenta que permite editar o material genético com precisão e atuar como sensor de trechos específicos de DNA, que vários laboratórios brasileiros já começaram a usar (ver Pesquisa FAPESP nº 240). Essas são algumas das ferramentas da biologia sintética, que pode prescindir dos fagos e montar sequências de DNA de forma completamente independente de organismos e assim construir bactérias com genoma planejado, uma possibilidade inaugurada em 2010 pelo bioquímico norte-americano Craig Venter (ver Pesquisa FAPESP nº 172).
Esse tipo de abordagem é incipiente no Brasil, mas já despontam projetos de modificação de microrganismos para produção de substâncias. “A potencial aplicação está no radar de todos aqueles que fazem biologia molecular com fins diagnósticos e para tratamento de doenças”, diz o médico Roger Chammas, da Faculdade de Medicina (FM) da USP.
A bióloga Aparecida Maria Fontes, da Faculdade de Medicina de Ribeirão Preto (FMRP) da USP, teve contato com a biologia sintética por ocasião de uma parceria com Venter. Junto com colegas, ela busca uma alternativa para o tratamento da doença de Gaucher, cujos portadores são deficientes em uma enzima que degrada um tipo de gordura. O acúmulo dessa gordura nas células causa uma série de problemas, até a morte. “Há cerca de 600 portadores no Brasil e o Ministério da Saúde gasta R$ 200 mil por ano para tratar cada um deles”, explica. Em colaboração com o norte-americano Ron Weiss, do Massachusetts Institute of Technology (MIT), ela selecionou duas moléculas a serem inseridas em vírus, que por sua vez levam o gene de interesse para o núcleo de células humanas. Resultados preliminares são animadores com linhagens de células in vitro. “Conseguimos produzir vírus estáveis, a molécula é funcional, a enzima é capaz de degradar o substrato e as células mantêm uma produção permanente da enzima”, afirma. Ela pretende submeter o pedido de patente ainda este ano.
 SOONHEE MOON / UNIVERSIDADE COLUMBIAOutros organismos também podem ser transformados em microfábricas por meio da biologia sintética. O grupo liderado pelo bioquímico Cleslei Zanelli, da Faculdade de Ciências Farmacêuticas da Universidade Estadual Paulista (Unesp) em Araraquara, busca miniaturizar e tornar mais eficiente a produção de substância ativa da espinheira-santa (Maytenus ilicifolia), usada na medicina popular para o tratamento de problemas gástricos. Para isso, os pesquisadores identificaram o gene responsável pela produção da enzima que sintetiza o fármaco, a friedelina, e o transferiram para leveduras Saccharomyces cerevisiae, conforme artigo de 2016 na Scientific Reports. “Estamos com o projeto em andamento para aumentar os níveis de produção para igual ou maior do que vemos na planta”, diz Zanelli. A vantagem da produção por leveduras é que, além de exigir muito menos espaço do que plantas, a produção pode ser constante – sem depender do crescimento ou de estações do ano. “Basta acrescentar açúcar, vitaminas e uma fonte de nitrogênio no meio de cultura, que elas sintetizam a substância o tempo todo”, conta. Uma vez otimizada a produção, o grupo de Araraquara, que inclui a química Maysa Furlan, pretende utilizar o sistema de levedura modificada para produzir outras substâncias de ação antitumoral e anti-inflamatória que já estão em testes clínicos.
SOONHEE MOON / UNIVERSIDADE COLUMBIAOutros organismos também podem ser transformados em microfábricas por meio da biologia sintética. O grupo liderado pelo bioquímico Cleslei Zanelli, da Faculdade de Ciências Farmacêuticas da Universidade Estadual Paulista (Unesp) em Araraquara, busca miniaturizar e tornar mais eficiente a produção de substância ativa da espinheira-santa (Maytenus ilicifolia), usada na medicina popular para o tratamento de problemas gástricos. Para isso, os pesquisadores identificaram o gene responsável pela produção da enzima que sintetiza o fármaco, a friedelina, e o transferiram para leveduras Saccharomyces cerevisiae, conforme artigo de 2016 na Scientific Reports. “Estamos com o projeto em andamento para aumentar os níveis de produção para igual ou maior do que vemos na planta”, diz Zanelli. A vantagem da produção por leveduras é que, além de exigir muito menos espaço do que plantas, a produção pode ser constante – sem depender do crescimento ou de estações do ano. “Basta acrescentar açúcar, vitaminas e uma fonte de nitrogênio no meio de cultura, que elas sintetizam a substância o tempo todo”, conta. Uma vez otimizada a produção, o grupo de Araraquara, que inclui a química Maysa Furlan, pretende utilizar o sistema de levedura modificada para produzir outras substâncias de ação antitumoral e anti-inflamatória que já estão em testes clínicos.
Algumas bactérias já são usadas na indústria para produzir compostos, como vitaminas e aminoácidos. O grupo da bióloga Danielle Pedrolli, também da Unesp de Araraquara, está construindo RNAs sintéticos para melhorar a capacidade da bactéria Bacillus subtilis de produzir vitamina B2 e suas precursoras, as purinas, por meio da ativação de cinco alvos no material genético. “Vamos conectar a ferramenta de RNA a um sistema que faz com que ela entre em ação quando a densidade de bactérias é adequada”, explica. Já funcionou in vitro, agora falta testar nas bactérias.
Essa capacidade das bactérias de detectar a densidade de companheiras tem usos importantes, como no laboratório do biólogo sintético norte-americano Tal Danino, da Universidade Columbia, em Nova York, que esteve em São Paulo em março para um evento científico. “Nosso laboratório busca maneiras de programar bactérias para detectar e tratar câncer”, afirma. “Desenhamos essas bactérias para se comunicarem, sincronizando seu ataque ao tumor com a produção de um fármaco.” O grupo programa sequências de DNA como códigos de computador e assim constrói circuitos que permitem, por exemplo, que as bactérias produzam substâncias tóxicas às células. “Podemos determinar que as bactérias expressem um gene apenas quando encontram um tumor”, conta o engenheiro biomédico Tetsuhiro Harimoto, estudante de doutorado no laboratório de Danino. Essa ação dirigida seria possível porque o ambiente dentro de um tumor, em termos de teor de oxigênio e de pH, por exemplo, é específico e pode ser programado como ideal para a atividade da bactéria sintética.
 Quando atingem uma densidade clínica dentro do tumor, as bactérias podem se romper e liberar o fármaco que produzem por engenharia genética. Além de direcionada com precisão, a medicação também se retroalimentaria. “Algumas bactérias sobrevivem e voltam a se multiplicar, formando um ciclo de crescimento e lise”, conta Harimoto. O grupo já teve bons resultados em células humanas in vitro e conseguiu reduzir tumores em camundongos, quando em combinação com quimioterapia, conforme descrito em artigo de 2016 na Nature.
Quando atingem uma densidade clínica dentro do tumor, as bactérias podem se romper e liberar o fármaco que produzem por engenharia genética. Além de direcionada com precisão, a medicação também se retroalimentaria. “Algumas bactérias sobrevivem e voltam a se multiplicar, formando um ciclo de crescimento e lise”, conta Harimoto. O grupo já teve bons resultados em células humanas in vitro e conseguiu reduzir tumores em camundongos, quando em combinação com quimioterapia, conforme descrito em artigo de 2016 na Nature.
Os resultados promissores explicam o otimismo de Danino, que prevê frutos terapêuticos em breve. “Há várias empresas e laboratórios, inclusive o meu, avançando na ideia de usar bactérias ou células desenhadas para doenças como câncer, diabetes e distúrbios intestinais”, conta. “É só uma questão de tempo até que melhoremos nossa engenharia desses organismos para que tenham um grande impacto na sociedade.”
Enquanto isso, ele também aplica a manipulação de bactérias para a arte, incluindo um projeto com o artista plástico brasileiro Vik Muniz. Para o pesquisador, o exercício estético pode ajudar a comunicar conceitos científicos complexos. “As artes visuais não apenas são universais, transcendendo barreiras de idioma e de jargão científico, elas convidam uma audiência mais ampla a ver a ciência e fazer perguntas sobre ela.”
Computação viva
O biólogo Rafael Silva Rocha, da FMRP-USP, investiga o funcionamento genético de bactérias usando um enfoque mais de computação do que de biologia molecular. “Temos desenvolvido pesquisa na área de reprogramação de bactérias, engenharia de fungos para bioenergia, desenvolvimento de biossensores e de ferramentas computacionais para engenharia de circuitos gênicos”, resume ele, que trabalha em parceria com sua mulher, a também bióloga María Eugenia Guazzaroni, professora no Departamento de Biologia da Faculdade de Filosofia, Ciências e Letras da mesma universidade.
 SOONHEE MOON / UNIVERSIDADE COLUMBIAEle exemplifica o trabalho com a estrutura genética da bactéria Escherichia coli: são 4.500 genes cujo funcionamento é orquestrado por 200 reguladores que agem de forma combinatória. Um gene pode ser controlado por 20 reguladores diferentes, enquanto um regulador pode afetar o funcionamento de centenas de genes. Ao estudar essas redes de interação, é possível selecionar os reguladores de ação mais ampla e usá-los para modular e alterar aspectos específicos do comportamento das bactérias. O grupo já produziu trabalhos iniciais, como o publicado em 2015 na ACS Synthetic Biology em que conseguiram inserir em bactérias vivas dois trechos sintéticos de DNA que regulam a atividade gênica. A ideia é integrar a informação sobre como o genoma das bactérias é regulado para implementar inovações biotecnológicas.
SOONHEE MOON / UNIVERSIDADE COLUMBIAEle exemplifica o trabalho com a estrutura genética da bactéria Escherichia coli: são 4.500 genes cujo funcionamento é orquestrado por 200 reguladores que agem de forma combinatória. Um gene pode ser controlado por 20 reguladores diferentes, enquanto um regulador pode afetar o funcionamento de centenas de genes. Ao estudar essas redes de interação, é possível selecionar os reguladores de ação mais ampla e usá-los para modular e alterar aspectos específicos do comportamento das bactérias. O grupo já produziu trabalhos iniciais, como o publicado em 2015 na ACS Synthetic Biology em que conseguiram inserir em bactérias vivas dois trechos sintéticos de DNA que regulam a atividade gênica. A ideia é integrar a informação sobre como o genoma das bactérias é regulado para implementar inovações biotecnológicas.
Essa integração de como funcionam as diferentes escalas da genética de um organismo – genômica, transcritômica, proteômica e o funcionamento de enzimas – também é um dos focos do grupo liderado pelo botânico Marcos Buckeridge, do Instituto de Biociências da USP. A bioinformata Amanda Rusiska Piovezani, sua aluna de doutorado, desenvolveu uma ferramenta computacional que permite explorar as relações entre os elementos da rede complexa que compõe o funcionamento de uma parte específica da raiz de plantas de cana-de-açúcar. Pode parecer um detalhe, mas o intuito é entender como se dá a formação de cavidades de ar (um tecido conhecido como aerênquima) nas raízes por meio da degradação da parede celular, uma propriedade essencial à otimização da produção de biocombustível. O software será integrado a uma ferramenta de teste estatístico desenvolvido pelo mestrando Vinícius Carvalho e poderá ser aplicado a uma variedade de outros sistemas. “Até ao funcionamento de uma cidade”, imagina Buckeridge. Ele está envolvido na criação de um centro de pesquisa chamado Sistemas de Biomassa e Biologia Sintética, a ser sediado na USP, que se concentrará justamente nesse tipo de abordagem para resolver problemas ligados à bioenergia, a partir do Programa Bioen da FAPESP.
Comunidade secreta
Há oito anos pesquisadores se debruçam sobre um objeto aparentemente insalubre: dejetos em decomposição dos animais do zoológico paulistano. Mas, para além do que se vê, a vida invisível das composteiras tem revelado uma riqueza importante. “Queremos fazer uma coleção do consórcio de bactérias e identificar aquelas promissoras para aplicações biotecnológicas”, conta o bioinformata João Setubal, do Instituto de Química da USP (IQ-USP).
Seu grupo extrai o DNA dessas amostras de composto em busca dos microrganismos que ali vivem e recentemente obteve o genoma quase completo de seis bactérias – quatro delas novas para a ciência, de acordo com artigo de abril na revista Frontiers in Microbiology. O composto também foi acompanhado por 100 dias, com coleta do material genético a cada 10 dias. “Queríamos ver como as populações microbianas variam ao longo do tempo”, explica Setubal. Um artigo publicado em dezembro na Scientific Reports mostra como a composição bacteriana vai mudando de acordo com a disponibilidade de nutrientes. As bactérias que vivem de celulose são mais abundantes no início, degradando essa substância. A lignina, que confere rigidez aos tecidos vegetais, é a última a ser degradada por microrganismos especializados. As enzimas produzidas por eles, estáveis nas altas temperaturas da composteira, são promissoras para aplicações industriais.
Artigos científicos
LEMOS, L. N. et al. Genome-centric analysis of a thermophilic and cellulolytic bacterial consortium derived from composting. Frontiers in Microbiology. v. 8, 644. 19 abr. 2017.
ANTUNES, L. P. et al. Microbial community structure and dynamics in thermophilic composting viewed through metagenomics and metatransciptomics. Scientific Reports. v. 6, 38915. 12 dez. 2016.
Competição movimenta biologia sintética
O Brasil está ganhando tradição em uma competição internacional que busca o avanço da biologia sintética por meio de projetos desenvolvidos por estudantes de graduação, o International Genetically Engineered Machine Competition (iGEM) (ver Pesquisa FAPESP nº 247). Este ano competem quatro grupos brasileiros: da Unesp de Araraquara, da Universidade Federal do Amazonas e duas unidades da USP – o campus da capital e a Escola Superior de Agricultura Luiz de Queiroz (Esalq), em Piracicaba. “É como se houvesse um catálogo com 2 mil peças de lego que podem ser combinadas para formar um circuito”, compara Rafael Silva Rocha, um dos coordenadores da equipe de São Paulo. “O objetivo não é chegar no produto final, mas avançar o máximo possível.” Um dos limites é o prazo de um ano, mas os projetos podem continuar a ser desenvolvidos após a competição, pelo mesmo grupo ou outros.
No início do ano, o Clube de Biologia Sintética da USP, ou SynbioBrasil, organizou um encontro com participantes de todo o Brasil. “Estamos mostrando para estudantes de outros lugares que vale a pena organizar clubes e que o iGEM abre portas”, conta o químico Otto Heringer, que acaba de concluir a graduação em química na USP e abriu uma empresa que pretende usar ferramentas de biologia sintética para combater pragas na agricultura. Ele se refere tanto ao aprendizado de como desenvolver um projeto na área como à rede social e científica que se cria em consequência.
O biólogo Cauã Westmann, estudante de mestrado no laboratório de Rocha, coleciona medalhas de prata do iGEM. Foram quatro em suas participações entre 2012 e 2016, uma delas como integrante do time da Universidade de York, no Reino Unido. Este ano ele está ajudando a organizar o trabalho da equipe paulistana da USP. “O Brasil tem uma situação única, com uma comunidade que está crescendo e se tornando muito colaborativa”, conta ele, que está organizando um clube de biologia sintética no campus de Ribeirão Preto da USP, nos moldes dos outros: um grupo de estudantes auto-organizado, sem a liderança de professores, que se encontra para discutir ciência. “É muito gratificante conseguir unir matemática, química, biologia, engenharia e até ciências sociais”, conta. Uma cientista social está estudando, em seu mestrado, o encontro científico proporcionado pelo Synbio. “O clube virou até tema de estudo!”
Projetos
1. Estudos da diversidade microbiana no Parque Zoológico do Estado de São Paulo (nº 11/50870-6); Modalidade Projeto Temático; Programa Biota; Pesquisador responsável João Carlos Setubal (USP); Investimento R$ 2.355.648,35.
2. Micobactérias e seus elementos extracromossômicos: Caracterização molecular e aplicações biotecnológicas (nº 11/18326-4); Modalidade Projeto Temático; Pesquisadora responsável Sylvia Luisa Pincherle Cardoso Leão (Unifesp); Investimento R$ 1.181.733,92.
3. PneumoPhageColor – desenvolvimento de um kit de biodetecção colorimétrica de Pseudomonas aeruginosa com base em partículas fágicas (nº 16/08884-3); Modalidade Auxílio à Pesquisa – Regular; Pesquisador responsável Vitor Manuel Cardoso Figueiredo Balcão (Uniso); Investimento R$ 138.199,67.
4. Abordagens de biologia sintética para decifrar os mecanismos de integração de sinais em promotores bacterianos complexos (nº 12/22921-8); Modalidade Jovem Pesquisador; Pesquisador responsável Rafael Silva Rocha (USP); Investimento R$ 1.370.671,34.
5. Clonagem e caracterização funcional das oxidoesqualeno ciclases de Maytenus ilicifolia (nº 14/03819-3); Modalidade Auxílio à Pesquisa – Regular; Pesquisador responsável Cleslei Fernando Zanelli (Unesp); Investimento R$ 235.403,07.
6. Synthetic biology, use of humanized codons and microRNAs for the production of a biopharmaceutical for Gaucher disease (nº 13/50450-2); Modalidade Auxílio à Pesquisa – Parceria para Inovação Tecnológica (Pite); Convênio Agilent; Pesquisadora responsável Aparecida Maria Fontes (USP); Investimento R$ 948.505,23.
7. Reprogramação do metabolismo de purina em Bacillus subtilis através de tecnologia de sRNA (nº 14/17564-7); Modalidade Jovem Pesquisador; Pesquisadora responsável Danielle Biscaro Pedrolli (Unesp); Investimento R$ 854.252,72.
8. Uso da abordagem de biologia de sistemas para desenvolver um modelo de funcionamento em plantas (nº 11/52065-3); Modalidade Auxílio à Pesquisa – Parceria para Inovação Tecnológica (Pite); Convênio Microsoft; Pesquisador responsável Marcos Silveira Buckeridge (USP); Investimento R$ 228.500,00 e US$ 12.000,00.
Artigos científicos
AMGARTEN, D. et al. Three novel Pseudomonas phages isolated from composting provide insights into the evolution and diversity of tailed phages. BMC Genomics. v. 18, n. 1, 346. 4 mai. 2017.
WATERS, E. M. et al. Phage therapy is highly effective against chronic lung infections with Pseudomonas aeruginosa. Thorax. v. 72, n. 7, p. 666-7. jul. 2017.
LIMA-JUNIOR, J. D. et al. Characterization of mycobacteria and mycobacteriophages isolated from compost at the São Paulo Zoo Park Foundation in Brazil and creation of the new mycobacteriophage Cluster U. BMC Microbiology. v. 16, n. 1, 111. 17 jun. 2016.
BALCÃO, V. M. et al. Structural and functional stabilization of phage particles in carbohydrate matrices for bacterial biosensing. Enzyme and Microbial Technology. v. 53, n. 1, p. 55-69. 10 jun. 2013.
BALCÃO, V. M. et al. Carbohydrate hydrogels with stabilized phage particles for Bacterial biosensing: bacterium diffusion studies. Applied Biochemistry and Biotechnology. v. 172, n. 3, p. 1194-214. fev. 2014.
AMORES, G. R. et al. Engineering synthetic cis-regulatory elements for simultaneous recognition of three transcriptional factors in bacteria. ACS Synthetic Biology. v. 4, n. 12, p. 1287-94. 18 dez. 2015.
DIN, M. O. et al. Synchronized cycles of bacterial lysis for in vivo delivery. Nature. v. 536, n. 7614, p. 81-5. 4 ago. 2016.
SOUZA-MOREIRA, T. M. et al. Friedelin Synthase from Maytenus ilicifolia: Leucine 482 Plays an essential role in the production of the most rearranged pentacyclic triterpene. Scientific Reports. v. 6, 36858. 22 nov. 2016.