Virus para combatir infecciones humanas, bacterias contra el cáncer, ADN programado como un código de computación. Son soluciones que busca la ciencia y que la evolución ya habría desarrollado en organismos unicelulares. Una de ellas es la existencia de virus capaces de identificar y destruir bacterias específicas. Se trata de los bacteriófagos, o simplemente fagos, cada vez más reconocidos como una posible alternativa a los antibióticos y fuente de aplicaciones biotecnológicas innovadoras. Otro frente de investigación, la biología sintética, manipula el material genético de los microorganismos y organiza a bacterias y levaduras para producir nuevos tratamientos, funcionar como sensores y abrir otras grandes –aunque microscópicas– posibilidades.
Muchos de esos proyectos surgen de la investigación básica, que apunta a la comprensión de organismos y mecanismos sin tener en vista inicialmente aplicaciones o productos. Éste es el caso del estudio microbiológico de las composteras del Parque Zoológico de São Paulo (lea en Pesquisa FAPESP edición nº 181), cuyo objetivo consistía en catalogar la diversidad bacteriana y estudiar los procesos de descomposición. Con todo, “allí donde proliferan las bacterias también hay muchos fagos”, dice la bioquímica Aline Maria da Silva, del Instituto de Química de la Universidad de São Paulo (IQ-USP), quien trabaja en el proyecto en colaboración con el bioinformático João Carlos Setubal, del mismo instituto. “Los fagos regulan las poblaciones de bacterias”, explica la investigadora, que al comienzo se interesó por los virus únicamente porque interferían en su objeto de estudio. Pero un alumno suyo a quien dirigía en su maestría, Deyvid Amgarten, un enamorado de los fagos, fue más allá y comenzó a analizar el material genético viral de las composteras que acumulan alimentos y detritos de los animales del zoológico.

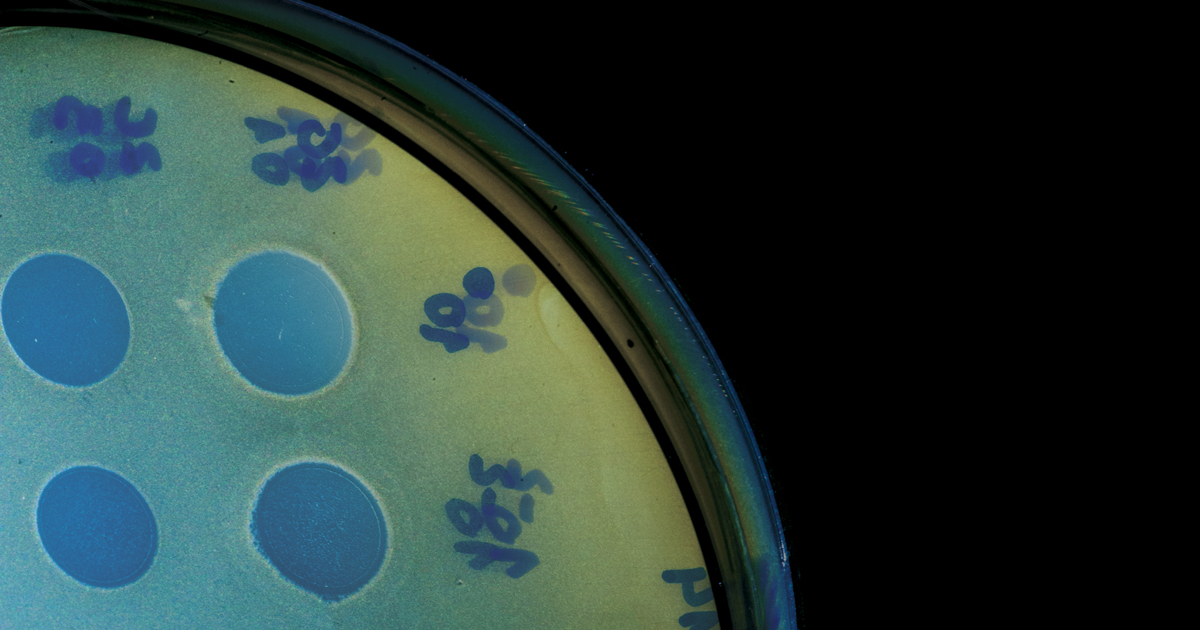
Victor Balcão/ Uniso
Los fagos más claros ya inocularon ADN en un fragmento de la bacteriaVictor Balcão/ UnisoComo una idea conduce a otra y despierta la curiosidad, el grupo se abocó a la búsqueda de bacteriófagos útiles para el combate contra las bacterias patógenas que causan enfermedades. En el compostaje, se hallaron bacterias del género Pseudomonas, parientes de la peligrosa P. aeruginosa, causante de infecciones hospitalarias. En un informe publicado a comienzos de año, la Organización Mundial de la Salud clasificó a esa bacteria como la segunda en términos de urgencia de estudios de nuevos antibióticos, frente a su alta resistencia a los fármacos existentes. La ventaja de los fagos reside en que evolucionan a la par de sus blancos, manteniendo su capacidad de ataque específico sobre un tipo de bacteria sin perjudicar al organismo humano.
El grupo del IQ consiguió aislar –y caracterizar genéticamente– tres bacteriófagos distintos, tal como se describe en un artículo publicado en el mes de mayo en la revista BMC Genomics. “Los fagos degradan una biopelícula de bacterias en 24 horas”, afirma Da Silva. Para saber cómo se comportan esos virus en el medio ambiente y en el interior del organismo de los mamíferos, sería necesario que otros grupos de investigación científica estuvieran interesados en la realización de esos test. Ya se han testeado con éxito otros fagos contra P. aeruginosa en ratones, según muestra un estudio conducido por el inmunólogo Aras Kadioglu, de la Universidad de Liverpool, en el Reino Unido, que fuera publicado en la edición del mes de julio de la revista Thorax. Ese tratamiento eliminó bacterias de los pulmones en un 70% de ratones con una infección afianzada, condición que entre los seres humanos es una de las causas principales de mortalidad hospitalaria.
El método para aislar los fagos en el IQ-USP es el mismo que se utiliza en aplicaciones médicas en el este de Europa. Desde 1923, el Instituto Eliava, en Tbilisi, Georgia, emplea agentes virales contra cualquier infección bacteriana, incluso en el caso de aquéllas que ningún otro tratamiento pudo eliminar (vea la infografía). Una revisión publicada en 2001 en la revista Antimicrobial Agents and Chemotherapy, realizada por investigadores del Eliava y de la Universidad de Maryland, en Estados Unidos, compara el éxito del tratamiento con antibióticos (un 64%) y fagos (un 82%), y afirma que la terapia viral es incluso más eficaz (un 95%) cuando se aplica por vía intravenosa. Por eso es que los consultan pacientes de todo el mundo. El relato histórico refiere que el descubrimiento de la posibilidad de utilizar virus como tratamiento fue concomitante con el de los antibióticos. Como la acción de estos es más amplia, y por eso más proclive para su producción industrial, esa estrategia cobró preponderancia mundial, excepto en el antiguo bloque soviético, donde el aislamiento impuesto por la cortina de hierro exigía soluciones creativas e independientes de lo que ocurría en Occidente.
A lo largo de las décadas de uso de antibióticos, la evolución trató de seleccionar bacterias resistentes dando origen a linajes responsables de graves casos de infecciones hospitalarias. A fines del mes de abril fue noticia el caso del psiquiatra estadounidense Tom Patterson, de la Facultad de Medicina de la Universidad de California en San Diego, quien al comienzo de 2016 entró en coma a causa de una infección que no respondía a los antibióticos. Su esposa, Steffanie Strathdee, y el médico Robert Schooley, ambos docentes en la misma facultad, apelaron a un último recurso y consiguieron autorización para intentar la terapia con fagos. “Se enviaron muestras del paciente a dos grupos de investigación que seleccionaron los fagos”, relata Schooley, quien aboga para que los bacteriófagos sean ampliamente reconocidos como parte del arsenal contra las infecciones bacterianas. “Empresas de varios países están comenzando a trabajar en el desarrollo de este tipo de terapia”, informa.
 Patterson salió del coma y su caso, que se está analizando publicarlo en un periódico científico, fue presentado el pasado 27 de abril en el Instituto Pasteur de París, en el marco del evento conmemorativo del centenario de la investigación con bacteriófagos. La fecha conmemora el descubrimiento de los fagos por el microbiólogo francocanadiense Felix d’Herelle (1873-1949), del Instituto Pasteur, en la misma época en que lo hiciera el británico Frederick Twort (1877-1950), y por eso se la bautizó como el Día de la Terapia con Fagos Humanos. Por cierto, D’Herelle fue partícipe de la fundación del Instituto Eliava, pero no regresó a Tbilisi luego de la ejecución del bacteriólogo George Eliava (1892-1937) por el régimen estalinista.
Patterson salió del coma y su caso, que se está analizando publicarlo en un periódico científico, fue presentado el pasado 27 de abril en el Instituto Pasteur de París, en el marco del evento conmemorativo del centenario de la investigación con bacteriófagos. La fecha conmemora el descubrimiento de los fagos por el microbiólogo francocanadiense Felix d’Herelle (1873-1949), del Instituto Pasteur, en la misma época en que lo hiciera el británico Frederick Twort (1877-1950), y por eso se la bautizó como el Día de la Terapia con Fagos Humanos. Por cierto, D’Herelle fue partícipe de la fundación del Instituto Eliava, pero no regresó a Tbilisi luego de la ejecución del bacteriólogo George Eliava (1892-1937) por el régimen estalinista.
Mientras que en la mayoría de los países la posibilidad de tratamiento se circunscribe a aquellos casos que se consideran perdidos, los científicos se limitan a caracterizar a esos abundantes y ubicuos virus. La microbióloga Sylvia Cardoso Leão, de la Universidad Federal de São Paulo (Unifesp), una experta en micobacterias –las causantes de la tuberculosis y otras infecciones de tratamiento difícil–, se interesó recientemente por los fagos. En colaboración con la microbióloga Cristina Viana Niero, del campus de Diadema de la Unifesp y participante del proyecto del zoológico. Cardoso Leão y su entonces alumno de maestría James Daltro Lima-Junior buscaron en las composteras fagos especializados en las micobacterias. Los hallaron, pero no a las bacterias con las cuales interactúan. En colaboración con Da Silva y Setubal, el grupo caracterizó a esos virus desde el punto de vista genético, y el biólogo molecular Graham Hatfull, de la Universidad de Pittsburgh, en Estados Unidos, comparó esa caracterización con las secuencias del archivo que mantiene. “Se trata de la mayor colección mundial de bacteriófagos que infectan micobacterias, una herramienta importante para entender mejor a esas especies” afirma Cardoso Leão. Lo sorprendente fue comprobar que uno de los fagos paulistanos estudiados es muy parecido a otro aislado de una muestra recabada en Sudáfrica. Acaso sea un aporte de algún animal africano residente en el zoológico.
Cardoso Leão monitorea infecciones por micobacterias y ayudó a establecer protocolos para combatir brotes y detectar la causa del problema, como en el caso de la resistencia a los desinfectantes que se usan en los hospitales. La investigadora sostiene que los fagos constituirían un buen aliado en esa batalla permanente. “Ellos son de lo más común, un arma de la naturaleza”. En la actualidad, pueden adquirirse productos a base de fagos para eliminar bacterias en verduras y disminuir el porcentaje de Salmonella en el pollo, por ejemplo, pero no para combatir enfermedades humanas.
 La capacidad de los fagos para reconocer sus bacterias correspondientes también puede dar origen a sensores, como los que el ingeniero de alimentos portugués Victor Balcão, de la Universidad de Sorocaba (Uniso), en el interior paulista, pretende elaborar en colaboración con la bioquímica Marta Vila, de la misma institución. “La idea consiste en la creación de una cinta adhesiva que pueda pegarse en las paredes de los hospitales para detectar Pseudomonas aeruginosa”, explica. El proyecto, al que se le dio el nombre de PneumoPhageColor, contempla revestir esos adhesivos con un gel atiborrado de fagos extraídos de las aguas cloacales de los hospitales. Si existieran bacterias en el aire, la idea es que las mismas atraviesen el hidrogel y su interacción con el virus y subsiguiente destrucción (un proceso al que se lo denomina lisis) produzca un brillo, tal como el que generan las luciérnagas y algunos hongos (lea en Pesquisa FAPESP, edición nº 255), por medio de una reacción química entre las sustancias integradas en el hidrogel. Esa señal de advertencia podría indicar la necesidad de realizar una desinfección en ese ambiente. “La probabilidad de entrar a un hospital y salir con una infección es de un 30%, advierte el investigador.
La capacidad de los fagos para reconocer sus bacterias correspondientes también puede dar origen a sensores, como los que el ingeniero de alimentos portugués Victor Balcão, de la Universidad de Sorocaba (Uniso), en el interior paulista, pretende elaborar en colaboración con la bioquímica Marta Vila, de la misma institución. “La idea consiste en la creación de una cinta adhesiva que pueda pegarse en las paredes de los hospitales para detectar Pseudomonas aeruginosa”, explica. El proyecto, al que se le dio el nombre de PneumoPhageColor, contempla revestir esos adhesivos con un gel atiborrado de fagos extraídos de las aguas cloacales de los hospitales. Si existieran bacterias en el aire, la idea es que las mismas atraviesen el hidrogel y su interacción con el virus y subsiguiente destrucción (un proceso al que se lo denomina lisis) produzca un brillo, tal como el que generan las luciérnagas y algunos hongos (lea en Pesquisa FAPESP, edición nº 255), por medio de una reacción química entre las sustancias integradas en el hidrogel. Esa señal de advertencia podría indicar la necesidad de realizar una desinfección en ese ambiente. “La probabilidad de entrar a un hospital y salir con una infección es de un 30%, advierte el investigador.
Esos son los pasos que éste se plantea ir dando hasta el final del año que viene, ahora que ha montado el laboratorio y que cuenta con la base de los logros obtenidos en el proyecto anterior. “Pudimos estabilizar el fago en el gel por confinamiento seguido de una reacción química”, relata, según lo que publicó en 2013 en la revista Enzyme and Microbial Technology. A continuación, el grupo de Sorocaba demostró que las bacterias tardan 6 minutos para penetrar 3 milímetros en el hidrogel, lo suficiente como para entrar en contacto con los fagos, tal como lo demostró en 2014 en la revista Applied Biochemistry and Biotechnology.

Soonhee Moon/ Universidad Columbia
Cultivos de bacterias teñidas para el estudio de su morfología se transforman en arte en el laboratorio de Tal DaninoSoonhee Moon/ Universidad ColumbiaControl genético
El panorama de las aplicaciones biotecnológicas con fagos es amplio y podría avanzar más allá de sacar provecho de sus habilidades naturales. Estos virus pueden alterarse genéticamente para actuar como sensores de patógenos o como armas contra ellos, o bien, para introducir secuencias genéticas deseadas al interior de las células. Otra propiedad de la interacción entre virus y bacterias es el sistema CRISPR-Cas9, una especie de sistema inmunológico de las bacterias que mantiene un registro de los virus con los que han tenido contacto. Ese sistema dio origen a una herramienta que permite editar el material genético con precisión y funcionar como sensor de tramos específicos del ADN, que varios laboratorios brasileños han comenzado a utilizar (lea en Pesquisa FAPESP, edición nº 240). Estas son algunas de las herramientas que aporta la biología sintética, que podría prescindir de los fagos y montar secuencias de ADN en forma completamente independiente de organismos y de ese modo generar bacterias con un genoma programado, una posibilidad inaugurada en 2010 por el bioquímico estadounidense Craig Venter (lea en Pesquisa FAPESP, edición nº 172).
Este tipo de abordaje es incipiente en Brasil, pero van apareciendo proyectos de modificación de microorganismos para la producción de sustancias. “Su aplicación potencial se encuentra bajo el radar de todos aquéllos que trabajan en biología molecular con fines diagnósticos y para el tratamiento de enfermedades”, dice el médico Roger Chammas, de la Facultad de Medicina de la USP (FM-USP).
La bióloga Aparecida Maria Fontes, de la Facultad de Medicina de Ribeirão Preto (FMRP) de la USP, tomó contacto con la biología sintética con ocasión de una colaboración que mantuvo con Venter. Junto a otros colegas, ella busca una alternativa para el tratamiento de la enfermedad de Gaucher, cuyos portadores padecen la deficiencia de un tipo de enzima que degrada una clase de lípidos. La acumulación de esa grasa en las células provoca una serie de problemas, y finalmente la muerte. “En Brasil hay alrededor de 600 portadores de la enfermedad y el Ministerio de Salud gasta 200 mil reales por año para el tratamiento de cada uno de ellos”, explica. En colaboración con el estadounidense Ron Weiss, del Massachusetts Institute of Technology (MIT), la investigadora seleccionó dos moléculas para insertarlas en virus que, a su vez, cargan con el gen de interés para el núcleo de células humanas. Los resultados preliminares son alentadores con los linajes de células in vitro. “Logramos producir virus estables, la molécula es funcional, la enzima es capaz de degradar el sustrato y las células mantienen una producción permanente de la enzima”, dice. Fontes pretende enviar la solicitud de patente este mismo año.
 Soonhee Moon/ Universidad ColumbiaOtros organismos también pueden ser transformados en microfábricas por medio de la biología sintética. El grupo encabezado por el bioquímico Cleslei Zanelli, de la Facultad de Ciencias Farmacéuticas de la Universidade Estadual Paulista (Unesp) en su campus de Araraquara, pretende miniaturizar y tornar más eficiente la producción del principio activo de la congorosa (Maytenus ilicifolia), que la medicina popular emplea para el tratamiento de problemas gástricos. Para ello, los investigadores identificaron al gen responsable de la producción de la enzima que sintetiza el fármaco, la friedelina, y lo transfirieron a levaduras Saccharomyces cerevisiae, tal como se describe en un artículo de 2016 en la revista Scientific Reports. “Tenemos un proyecto en curso para elevar los niveles de producción a una cantidad igual o mayor a lo que vemos en la planta”, dice Zanelli. La ventaja de la producción mediante levaduras radica en que además de requerir mucho menos espacio que las plantas, su producción puede ser constante, sin depender del crecimiento o de las estaciones del año. “Basta con agregarle azúcar, vitaminas y una fuente de nitrógeno al medio de cultivo, para que ésts sinteticen la sustancia en forma permanente”, comenta. Una vez optimizada la producción, el grupo de Araraquara, que incluye a la química Maysa Furlan, se propone emplear el sistema de levadura modificada para producir otras sustancias de acción antitumoral y antiinflamatoria que ya se encuentran en fase de ensayos clínicos.
Soonhee Moon/ Universidad ColumbiaOtros organismos también pueden ser transformados en microfábricas por medio de la biología sintética. El grupo encabezado por el bioquímico Cleslei Zanelli, de la Facultad de Ciencias Farmacéuticas de la Universidade Estadual Paulista (Unesp) en su campus de Araraquara, pretende miniaturizar y tornar más eficiente la producción del principio activo de la congorosa (Maytenus ilicifolia), que la medicina popular emplea para el tratamiento de problemas gástricos. Para ello, los investigadores identificaron al gen responsable de la producción de la enzima que sintetiza el fármaco, la friedelina, y lo transfirieron a levaduras Saccharomyces cerevisiae, tal como se describe en un artículo de 2016 en la revista Scientific Reports. “Tenemos un proyecto en curso para elevar los niveles de producción a una cantidad igual o mayor a lo que vemos en la planta”, dice Zanelli. La ventaja de la producción mediante levaduras radica en que además de requerir mucho menos espacio que las plantas, su producción puede ser constante, sin depender del crecimiento o de las estaciones del año. “Basta con agregarle azúcar, vitaminas y una fuente de nitrógeno al medio de cultivo, para que ésts sinteticen la sustancia en forma permanente”, comenta. Una vez optimizada la producción, el grupo de Araraquara, que incluye a la química Maysa Furlan, se propone emplear el sistema de levadura modificada para producir otras sustancias de acción antitumoral y antiinflamatoria que ya se encuentran en fase de ensayos clínicos.
La industria ya utiliza algunas bacterias para elaborar compuestos, tales como vitaminas y aminoácidos. El grupo de la bióloga Danielle Pedrolli, también de la Unesp de Araraquara, está fabricando ARNs sintéticos para mejorar la capacidad de producción de vitamina B2 y sus precursoras, las purinas, por la bacteria Bacillus subtilis, por medio de la activación de cinco objetivos en el material genético. “Conectaremos la herramienta de ARN a un sistema que hace que ella entre en acción cuando la densidad de bacterias es la adecuada”, explica. Esta técnica ya funcionó in vitro, ahora resta testearla en las bacterias.
Esa capacidad de las bacterias para detectar la densidad de otras homólogas tiene usos importantes, tal como ocurre en el laboratorio del biólogo sintético estadounidense Tal Danino, de la Universidad Columbia, en Nueva York, quien visitó São Paulo en el marco de un evento científico. “Nuestro laboratorio busca maneras de programar bacterias para detectar y tratar el cáncer”, afirma. “Diseñamos esas bacterias para que se comuniquen, sincronizando su ataque al tumor mediante la producción de un fármaco”. El grupo programa secuencias de ADN similares a códigos de computadora y de ese modo construye circuitos que permiten, por ejemplo, que las bacterias produzcan sustancias tóxicas para las células. “Podemos determinar que las bacterias expresen un gen solamente cuando detectan un tumor”, relata el ingeniero biomédico Tetsuhiro Harimoto, alumno de doctorado en el laboratorio de Danino. Esa actividad dirigida sería posible porque el ambiente en el interior de un tumor, en cuanto al nivel de oxígeno y a su pH, por ejemplo, es específico y puede programarse como algo ideal para la actividad de la bacteria sintética.
 Cuando alcanzan cierta densidad clínica dentro del tumor, las bacterias pueden romperse y liberar el fármaco que producen por ingeniería genética. Además de poder direccionarla con precisión, la medicación también se retroalimentaría. “Algunas bacterias sobreviven y vuelven a multiplicarse, instalando un ciclo de crecimiento y lisis”, comenta Harimoto. El grupo ha tenido buenos resultados en células humanas in vitro y logró reducir tumores en ratones cuando el tratamiento fue combinado con quimioterapia, según se ha descrito en un artículo de 2016 en la revista Nature.
Cuando alcanzan cierta densidad clínica dentro del tumor, las bacterias pueden romperse y liberar el fármaco que producen por ingeniería genética. Además de poder direccionarla con precisión, la medicación también se retroalimentaría. “Algunas bacterias sobreviven y vuelven a multiplicarse, instalando un ciclo de crecimiento y lisis”, comenta Harimoto. El grupo ha tenido buenos resultados en células humanas in vitro y logró reducir tumores en ratones cuando el tratamiento fue combinado con quimioterapia, según se ha descrito en un artículo de 2016 en la revista Nature.
Los resultados alentadores explican el optimismo de Danino, que anticipa frutos terapéuticos a corto plazo. “Hay varias empresas y laboratorios, el mío inclusive, que están avanzando en la idea de utilizar bacterias o células diseñadas para el tratamiento de enfermedades tales como el cáncer, diabetes y trastornos intestinales”, comenta. “Sólo es cuestión de tiempo hasta que logremos perfeccionar nuestra ingeniería de esos organismos para que alcancen un gran impacto en la sociedad”.
Mientras tanto, también lo aplica a la manipulación de bacterias en el arte, que incluye un proyecto junto al artista plástico brasileño Vik Muniz. Para el investigador, el ejercicio estético puede colaborar en la comunicación de conceptos científicos complejos. “Las artes visuales no sólo son universales: al trascender las barreras del idioma y de la jerga científica, ellas invitan a un público más amplio a observar la ciencia y a formular preguntas al respecto de ella”.
Computación viva
El biólogo Rafael Silva Rocha, de la FMRP-USP, estudia el funcionamiento genético de bacterias valiéndose de un enfoque más cercano a la computación que a la biología molecular. “Hemos desarrollado una investigación en el área de reprogramación de bacterias, ingeniería de hongos para bioenergía, desarrollo de biosensores y herramientas computacionales para ingeniería de circuitos génicos”, resume. Silva Rocha trabaja en forma conjunta con su esposa, María Eugenia Guazzaroni, también bióloga y docente del Departamento de Biología de la Facultad de Filosofía, Ciencias y Letras de la misma universidad.
 Soonhee Moon/ Universidad ColumbiaA modo de ejemplo, el investigador se refiere al trabajo con la estructura genética de la bacteria Escherichia coli: son 4.500 genes cuyo funcionamiento está orquestado por 200 reguladores que actúan en forma combinada. Un gen puede ser controlado por 20 reguladores diferentes, mientras que un regulador puede afectar el funcionamiento de cientos de genes. Al estudiar esas redes de interacción, pueden seleccionarse los reguladores de acción más amplia y utilizarlos para modular y alterar aspectos específicos del comportamiento de las bacterias. El grupo ha elaborado trabajos iniciales, tal como el que publicaron en 2015 en la revista ACS Synthetic Biology, en el cual lograron insertar en bacterias vivas dos tramos sintéticos de ADN que regulan la actividad génica. La idea consiste en integrar la información al respecto del modo en que se regula el genoma de las bacterias para implementar innovaciones biotecnológicas.
Soonhee Moon/ Universidad ColumbiaA modo de ejemplo, el investigador se refiere al trabajo con la estructura genética de la bacteria Escherichia coli: son 4.500 genes cuyo funcionamiento está orquestado por 200 reguladores que actúan en forma combinada. Un gen puede ser controlado por 20 reguladores diferentes, mientras que un regulador puede afectar el funcionamiento de cientos de genes. Al estudiar esas redes de interacción, pueden seleccionarse los reguladores de acción más amplia y utilizarlos para modular y alterar aspectos específicos del comportamiento de las bacterias. El grupo ha elaborado trabajos iniciales, tal como el que publicaron en 2015 en la revista ACS Synthetic Biology, en el cual lograron insertar en bacterias vivas dos tramos sintéticos de ADN que regulan la actividad génica. La idea consiste en integrar la información al respecto del modo en que se regula el genoma de las bacterias para implementar innovaciones biotecnológicas.
Esa integración de cómo funcionan las diferentes escalas de la genética de un organismo –genómica, transcriptómica, proteómica y el funcionamiento de enzimas– también es uno de los enfoques del grupo encabezado por el botánico Marcos Buckeridge, del Instituto de Biociencias de la USP. La bioinformática Amanda Rusiska Piovenzani, su alumna de doctorado, desarrolló una herramienta informática que permite explorar las relaciones entre los elementos de la red compleja que integra el funcionamiento de una parte específica de la raíz de las plantas de caña de azúcar. Podría parecer sólo un detalle, pero la meta es entender cómo se produce la formación de cavidades de aire (un tejido conocido con el nombre de aerénquima) en las raíces por medio de la degradación de la pared celular, una propiedad esencial para la optimización de la producción de biocombustible. El software estará integrado a una herramienta de test estadístico desarrollada por el maestrando Vinícius Carvalho y podría aplicarse a toda una gama de otros sistemas. “Incluso al funcionamiento de una ciudad”, imagina Buckeridge. El investigador se encuentra abocado a la creación de un centro de investigación denominado Sistemas de Biomasa y Biología Sintética, cuya sede funcionará en la USP, que se concentrará precisamente en ese tipo de abordaje para la resolución de problemas relacionados con la bioenergía, a partir del Programa Bioen de la FAPESP.

Eduardo Cesar
Investigadora recolecta material para la extracción de ADN en la compostera del zoológico, en 2011Eduardo CesarHace ocho años que los científicos vienen estudiando un objeto en apariencia insalubre: detritos en descomposición de los animales del zoológico paulistano. No obstante, más allá de lo que se ve, la vida invisible de las composteras ha revelado una riqueza importante. “Queremos armar un catálogo del consorcio de bacterias identificando a aquéllas que son prometedoras para aplicaciones biotecnológicas”, comenta el bioinformático João Setubal, del Instituto de Química de la USP (IQ-USP).
Su grupo de trabajo extrajo el ADN de esas muestras del compuesto en busca de los microorganismos que pululan allí y recientemente obtuvo el genoma casi completo de seis bacterias, cuatro de ellas nuevas para la ciencia, según se consigna en un artículo publicado en el mes de abril en la revista Frontiers in Microbiology. El compuesto también fue estudiado durante 100 días, con colecta del material genético cada 10 días. “Queríamos ver cómo varían las poblaciones microbianas a lo largo del tiempo”, explica Setubal. Un artículo publicado en diciembre en la revista Scientific Reports revela que la composición bacteriana va cambiando de acuerdo con la disponibilidad de nutrientes. Las bacterias que se nutren de celulosa son más abundantes al principio, degradando esa sustancia. La lignina, que les confiere rigidez a los tejidos vegetales, es la última en ser degradada por microorganismos especializados. Las enzimas que éstos producen, estables en las altas temperaturas de la compostera, son prometedoras para aplicaciones industriales.
Artículos científicos
LEMOS, L. N. et al. Genome-centric analysis of a thermophilic and cellulolytic bacterial consortium derived from composting. Frontiers in Microbiology. v. 8, 644. 19 abr. 2017.
ANTUNES, L. P. et al. Microbial community structure and dynamics in thermophilic composting viewed through metagenomics and metatransciptomics. Scientific Reports. v. 6, 38915. 12 dic. 2016.
Brasil está adquiriendo preeminencia en una competencia internacional que apunta al avance de la biología sintética por medio de proyectos desarrollados por estudiantes de carreras de grado, el International Genetically Engineered Machine Competition (iGEM) (lea en Pesquisa FAPESP, edición nº 247). Este año compiten cuatro equipos brasileños: de la Unesp de Araraquara, de la Universidad Federal de Amazonas y dos unidades de la USP, el campus que posee esta última en la capital y la Escuela Superior de Agricultura Luiz de Queiroz (Esalq), en Piracicaba. “Es como si contáramos con un catálogo con 2 mil piezas de lego que pueden combinarse para formar un circuito”, compara Rafael Silva Rocha, uno de los coordinadores del equipo de São Paulo. “La meta no consiste en llegar a un producto final, sino en avanzar lo máximo posible”. Uno de los límites es el plazo de un año, pero los proyectos pueden proseguir y ser desarrollados luego de la competencia por el mismo grupo u otros.
A comienzos de año, el Club de Biología Sintética de la USP, denominado SynbioBrasil, organizó un encuentro con participantes de todo Brasil. “Les estamos mostrando a los estudiantes de otros sitios que vale la pena armar clubes y que el iGEM abre puertas”, comenta el químico Otto Heringer, quien acaba de recibirse como químico en la USP y fundó una empresa que pretende usar herramientas de la biología sintética para combatir plagas en la agricultura. Heringer se refiere tanto al aprendizaje sobre el modo de desarrollar un proyecto en el área como a la red social y científica que se genera como consecuencia.
El biólogo Cauã Westmann, alumno de maestría en el laboratorio de Rocha, colecciona medallas de plata del iGEM. Fueron cuatro las que obtuvo en sus participaciones entre 2012 y 2016, una de ellas como integrante del equipo de la Universidad de York, en el Reino Unido. Este año está ayudando a organizar el trabajo del equipo paulistano de la USP. “Brasil dispone de una situación única, con una comunidad que está en crecimiento y tornándose muy colaborativa”, comenta el biólogo, quien está organizando un club de biología sintética en el campus de Ribeirão Preto de la USP, con el formato de los otros: un grupo de estudiantes autoorganizado, sin liderazgos docentes, que se encuentra para debatir sobre ciencia. “Resulta muy gratificante lograr aunar matemática, química, biología, ingeniería e incluso ciencias sociales”, comenta. Una socióloga está estudiando en su maestría el encuentro científico que genera el Synbio. “¡El club se ha transformado incluso en tema de estudio!”
Proyectos
1. Estudios de la diversidad microbiana en el Parque Zoológico del Estado de São Paulo (nº 11/50870-6); Modalidad Proyecto Temático; Programa Biota; Investigador responsable João Carlos Setubal (USP); Inversión R$ 2.355.648,35
2. Micobacterias y sus elementos extracromosómicos: Caracterización molecular y aplicaciones biotecnológicas (nº 11/18326-4); Modalidad Proyecto Temático; Investigadora responsable Sylvia Luisa Pincherle Cardoso Leão (Unifesp); Inversión R$ 1.181.733,92
3. PneumoPhageColor – desarrollo de un kit de biodetección colorimétrica de Pseudomonas aeruginosa con base en partículas fágicas (nº 16/08884-3); Modalidad Ayuda a la Investigación – Regular; Investigador responsable Vitor Manuel Cardoso Figueiredo Balcão (Uniso); Inversión R$ 138.199,67
4. Abordajes de biología sintética para descifrar los mecanismos de integración de señales en promotores bacterianos complejos (nº 12/22921-8); Modalidad Joven Investigador; Investigador responsable Rafael Silva Rocha (USP); Inversión R$ 1.370.671,34
5. Clonación y caracterización funcional de las oxidoescualeno ciclasas de Maytenus ilicifolia (nº 14/03819-3); Modalidad Ayuda a la Investigación – Regular; Investigador responsable Cleslei Fernando Zanelli (Unesp); Inversión R$ 235.403,07
6. Synthetic biology, use of humanized codons and microRNAs for the production of a biopharmaceutical for Gaucher disease (nº 13/50450-2); Modalidad Ayuda a la Investigación – Cooperación para la Innovación Tecnológica (Pite); Convenio Agilent; Investigadora responsable Aparecida Maria Fontes (USP); Inversión R$ 948.505,23
7. Reprogramación del metabolismo de la purina en Bacillus subtilis a través de la tecnología de sARN (nº 14/17564-7); Modalidad Joven Investigador; Investigadora responsable Danielle Biscaro Pedrolli (Unesp); Inversión R$ 854.252,72
8. Uso del abordaje de la biología de sistemas para el desarrollo de un modelo de funcionamiento en plantas (nº 11/52065-3); Modalidad Ayuda a la Investigación – Cooperación para la Innovación Tecnológica (Pite); Convenio Microsoft; Investigador responsable Marcos Silveira Buckeridge (USP); Inversión R$ 228.500,00 y US$ 12.000,00