Parecia festa de premiação do Oscar, com tapete vermelho, troféus e aplausos efusivos de uma plateia de mais de mil pessoas reunidas na imponente Sala São Paulo, na capital paulista. A premiação aos 193 pesquisadores de 35 laboratórios paulistas que concluíram o pioneiro sequenciamento genético de um fitopatógeno, a bactéria Xylella fastidiosa, eternizou a noite de 21 de fevereiro de 2000.
O estudo brasileiro foi a capa da Nature, uma das mais importantes revistas científicas do mundo, em 13 de julho de 2000. O editorial destacava que o Brasil desmentia a concepção de que apenas os países mais industrializados teriam potencial e pessoal qualificado para realizar pesquisa de ponta. Depois, jornais e revistas do Reino Unido, Estados Unidos e França noticiaram o sequenciamento da Xylella.

Leonardo CostaTroféu Árvore dos Enigmas, oferecido pelo governo do estado às instituições que coordenaram o trabalhoLeonardo Costa
“Ainda me lembro do barulho que o modem fazia”, brinca a bióloga Anamaria Aranha Camargo, do Centro de Oncologia Molecular do Hospital Sírio-libanês. Para ela, o mais importante foram a dedicação e a união dos grupos de trabalho, superando dificuldades de logística, em tempos de internet discada, e falta de experiência.
“A maioria dos pesquisadores não trabalhava diretamente com doença de plantas e teve de conciliar esse projeto entre suas prioridades”, ela conta. O hematologista Marco Antonio Zago, presidente do Conselho Superior da FAPESP, rememora: “Aquela foi a primeira vez que se mostrou a vantagem e a eficiência do trabalho em rede. Todos os laboratórios funcionavam como se fossem um só”.
Sem essa integração, não teria dado certo o plano do biólogo Fernando Reinach, então no Instituto de Química da Universidade de São Paulo e coordenador de área de bioquímica da FAPESP, a quem coube idealizar as bases do projeto (ver Pesquisa FAPESP nº 100). Em maio de 1997, o então diretor científico da FAPESP, o físico matemático José Fernando Perez, desafiou-o a criar um projeto de biotecnologia, alertando: “Estamos ficando para trás no campo da genética molecular”. Reinach pensou num objeto de pesquisa que desse a todos os laboratórios as mesmas oportunidades de capacitação. “Teria de ser um projeto no qual todo mundo pudesse aprender a mesma coisa, fazendo todas as etapas do trabalho. Daí surgiu a ideia de sequenciar um genoma, cada equipe sequenciando uma parte”, conta o biólogo. A conexão por meio da incipiente internet permitiria o trabalho de forma descentralizada, sem a necessidade de uma sede física.
Para a coordenação geral do projeto decidiu-se convidar Andrew Simpson, bioquímico britânico naturalizado brasileiro e então chefe do Laboratório de Genética do Instituto Ludwig de Pesquisa sobre o Câncer em São Paulo. Os laboratórios de Reinach na USP e do também biólogo Paulo Arruda na Universidade Estadual de Campinas (Unicamp) dariam apoio às equipes que enfrentassem alguma dificuldade. “Os laboratórios aprenderam fazendo”, diz Reinach.

ReproduçãoCapa da Nature de julho de 2000 com a descrição do genoma da bactéria causadora do amarelinhoReprodução
Nasce a Onsa
A fim de conferir respaldo científico ao trabalho, para o qual a FAPESP destinara US$ 12 milhões e o Fundo de Desenvolvimento da Citricultura (Fundecitrus) US$ 650 mil, Reinach convidou especialistas da França e do Reino Unido para acompanhar o trabalho. Mesmo assim, o projeto de sequenciamento fora do eixo Nova York-Paris-Londres era ousado. Perez alimentava essa ousadia: “Nossa cultura científica não estimula o risco”.
Assim, com ousadia e um toque de bom humor, criou-se a rede virtual de laboratórios Onsa, sigla para Organization for Nucleotide Sequencing and Analysis, ou Organização para Sequenciamento e Análise de Nucleotídeos. Foi uma brincadeira com The Institute for Genomic Research (TIGR, tigre, em inglês), a instituição norte-americana que, dois anos antes, sequenciara o primeiro genoma de uma bactéria de vida livre, Haemophilus influenzae.
O processamento dos dados gerados pelos laboratórios representou outro desafio – o genoma da bactéria era grande, com 2,7 milhões de pares de bases e 2.904 genes. “Os consultores do exterior alertaram que a bioinformática seria um gargalo no projeto. Aconselharam a contratação de um especialista europeu. Mas aí apareceram dois jovens, o matemático João Meidanis e o engenheiro mecânico João Setúbal, ambos da Unicamp, e disseram: ‘Vamos fazer a bioinformática, sabemos fazer’”, orgulha-se Perez.
“Eu havia trabalhado com bioinformática no doutorado nos Estados Unidos, sonhava que chegasse logo ao Brasil, mas achava que ainda ia demorar anos ou décadas”, conta Meidanis. “Começou mais cedo do que eu imaginava, fiquei maravilhado.”.
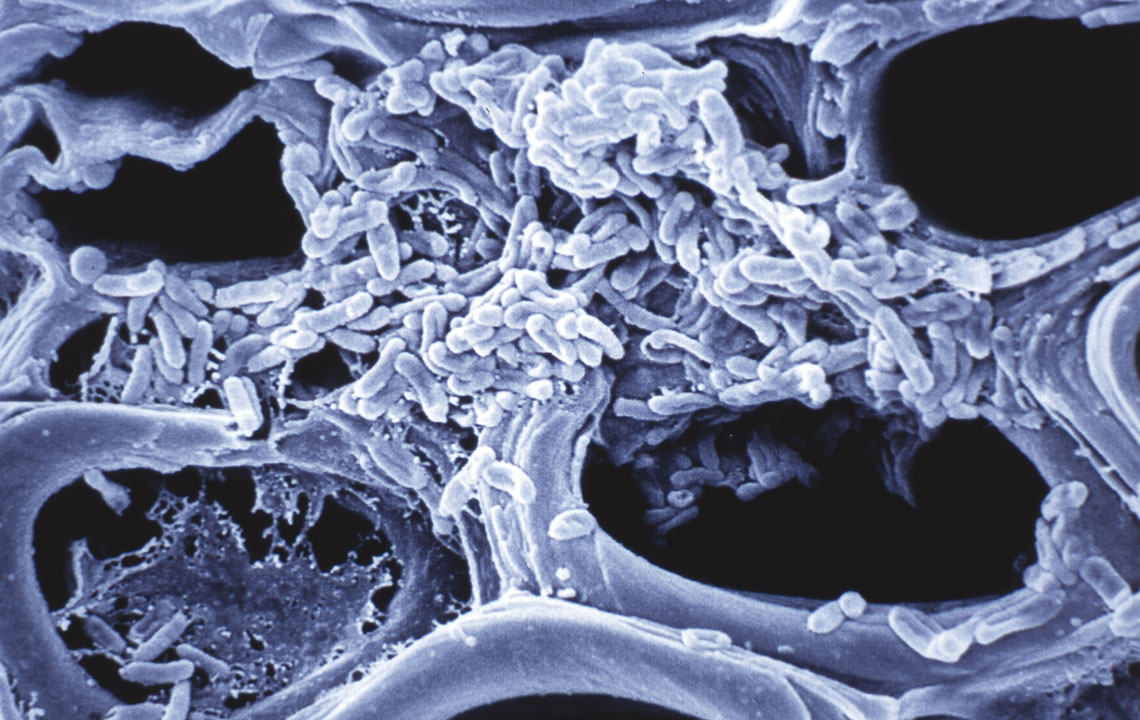
ELLOT W. KITAJIMA / USPImagem de microscopia dos biofilmes formados pela Xylella no interior das plantasELLOT W. KITAJIMA / USP
Houve, também, momentos de tensão. “Tínhamos um operador de TI [tecnologia da informação], que, como nós, era um experimentador”, conta Meidanis. “No meio do projeto, ele resolveu atualizar o sistema operacional da máquina principal. Só que deu errado. Ele tinha feito backup de tudo antes de começar, mas até restaurar os dados foram três dias de agonia.”
Em dois anos, o sequenciamento estava concluído. Para Perez, o sequenciamento do genoma foi apenas um dos resultados do projeto. Igualmente importantes foram, segundo ele, a capacitação de pesquisadores e laboratórios e o nascimento da bioinformática no Brasil.
A bactéria escolhida para ser sequenciada poderia nem ter sido Xylella fastidiosa. A ideia inicial era apenas que a escolha deveria recair sobre algo que não competisse com o que já era feito no exterior. Na seleção das candidatas, sobressaiu a sugestão do agrônomo Marcos Machado. Na época ele coordenava o Laboratório de Biotecnologia do Centro de Citricultura Sylvio Moreira, do Instituto Agronômico de Campinas (IAC) em Cordeirópolis, e tinha um ótimo argumento em favor da Xylella: era a causadora da Clorose Variegada dos Citros (CVC), também conhecida como amarelinho, praga que atingia 34% dos laranjais paulistas.

fundecitrusDetalhe do mapa genômico dos 2,7 milhões de pares de bases do cromossomo da X. fastidiosafundecitrus
“Não era uma bactéria fácil de trabalhar”, admite Machado. Dizia-se que o cientista francês Joseph-Marie Bové era o único que conseguia cultivar a Xylella em laboratório – foi o grupo dele que provou a relação causal entre a bactéria e a CVC. Machado tinha o contato dele e um histórico de parceria com a Universidade de Bordeaux. “Cultivamos a bactéria original enviada pelo Bové e entregamos o equivalente a 1 litro e meio de cultura ao Simpson, que providenciou a purificação do DNA”, lembra.
Para Simpson, o Projeto Genoma teve sucesso devido à soma de muitos talentos individuais articulados no trabalho em equipe: “O projeto inteiro foi a soma de muitos momentos de brilhantismo individual trabalhando junto”. Um problema que apareceu no final do sequenciamento revelou a capacidade da equipe. “Havia ficado um ‘buraco’ que ninguém sabia como fechar. Foi a percepção da Anamaria Camargo que permitiu que fechássemos o genoma”, conta Simpson. “Ela levantou a hipótese de que a bactéria tivesse duas cópias de RNA ribossomal [RNA armazenado em ribossomos, uma das estruturas celulares]. E fechou, resolveu”.

ReproduçãoUm dos efeitos do amarelinho: frutos menoresReprodução
“A sistematização das discussões de resultados científicos em grupo foi um grande aprendizado do projeto”, avalia a bióloga Marie-Anne Van Sluys, da Universidade de São Paulo. Ela conta que havia reuniões semanais e quinzenais nos grupos pequenos, reuniões de atualização a cada semestre e uma reunião anual com o comitê de avaliação. “Foi uma dinâmica que acabou sendo replicada em outros programas da FAPESP, como o Biota”, diz ela.
Herdeiros da Xylella
Assim que terminou o sequenciamento, em 1999, começou o Projeto Genoma Funcional da Xylella, focado nos genes relacionados à sua capacidade de causar doença nas plantas. O projeto reuniu 21 laboratórios e revelou os mecanismos de ação da bactéria, permitindo a criação de estratégias de controle, como poda de ramos infectados e produção de mudas em ambiente protegido do inseto-vetor, explica a bióloga Alessandra Alves de Souza, diretora de Pesquisa e Desenvolvimento do IAC, que trabalhou no projeto durante seu doutorado.

Miguel Boyayan / Revista Pesquisa FAPESPReunião de preparação do artigo científico com as conclusões do Projeto Genoma XylellaMiguel Boyayan / Revista Pesquisa FAPESP
Já sabendo que a bactéria obstruía o xilema, os vasos que distribuem água e sais minerais para a planta, ela descobriu uma molécula eficaz contra o patógeno: a N-acetilcisteína (NAC), apresentada em um artigo publicado em agosto de 2013 na PLOS ONE. A molécula estimulou a criação de uma startup, a CiaCamp, incubada no Centro de Citricultura do IAC.
Mais recentemente Souza desenvolveu uma variedade de laranjeira transgênica resistente à bactéria Xylella. Ela introduziu no genoma da planta um gene do patógeno, o rpfF, responsável pela produção de uma proteína que reduz sua movimentação. Ao reduzir a mobilidade da bactéria, impede-se que ela se espalhe pela planta.
A lista de herdeiros diretos do Projeto Genoma inclui o Genoma Humano do Câncer e diversos outros voltadosx ao campo da agronomia, como o Genoma da Cana-de-açúcar e de Xanthomonas citri, bactéria causadora do cancro cítrico (ver cronologia abaixo). Mesmo depois de encerrado o Programa Genoma, em 2008, a genômica continuou a receber atenção da FAPESP, que apoiou 262 projetos de pesquisa nessa área até novembro de 2025. “A ciência genômica que existe no Brasil começou na FAPESP com o projeto da Xylella”, reitera Zago. Ele destaca que o sequenciamento do vírus da Covid-19 – realizado em apenas dois dias após a confirmação do primeiro caso no Brasil, em fevereiro de 2020 – também é herdeiro desse projeto.

