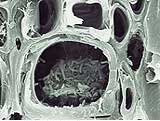
EDUARDO ALVES / ESALQ-USPColonias de Xylella creciendo dentro de los vasos conductores de savia: su acción comienza a ser contenidaEDUARDO ALVES / ESALQ-USP
La primera variedad de naranja dulce genéticamente modificada, que abre el camino para la producción de naranjos resistentes a las enfermedades, y una serie de bacterias mutantes, en las cuales fueron desactivados los genes considerados dañinos para las plantas, son algunas de las novedades del 1º Simposio Genoma Funcional de la Xylella fastidiosa, realizado entre los días 10 y 13 de diciembre en Serra Negra. Allí, los coordinadores de los 21 grupos de investigación del proyecto Genoma Funcional, financiado por la FAPESP, anunciaron las estrategias de combate a la Xylella fastidiosa, la bacteria causante de la enfermedad conocida como plaga amarilla o veteado cloroso cítrico (CVC, sigla en portugués).
Transmitida por un insecto – la cigarrita -, es una plaga que presenta síntomas más o menos graves, afecta a 65 millones de naranjos en el estado de São Paulo (un 36% del total) y transforma en improductivos a alrededor de seis millones de árboles por año. Los objetivos de los investigadores son claros: son los genes que permiten que la Xylella desencadene la enfermedad (le confieren patogenicidad) o determinan la agresividad (virulencia) con la que la planta será infectada.
El trabajo, iniciado hace dos años, converge ahora en la búsqueda de mecanismos – tales como plantas o bacterias alteradas genéticamente – que bloqueen la acción de dichos genes dañinos, y permitan el desarrollo de insecticidas más eficientes para impedir que la cigarrita les transmita la bacteria a plantas sanas o que lleven al desarrollo de plantas resistentes, productoras de proteínas capaces de impedir a supervivencia de la Xylella.
Se inicia de esta manera una nueva etapa en la lucha contra la bacteria causante de la plaga amarilla, una batalla que comenzó en el año 2000, con el exitoso secuenciamiento de su genoma en el marco de un programa financiado por la FAPESP. Más allá de las técnicas actualmente en uso – control de los insectos transmisores y uso de plantines exentos de contaminación, que al menos evitan la dispersión del problema entre plantas aún intactas -, será posible contar con los recursos de la ingeniería genética para construir bacterias o plantas modificadas genéticamente. Con ello se pretende bloquear el desarrollo de la plaga, que le ocasiona pérdidas anuales a la citricultura paulista estimadas en 100 millones de dólares.
En el simposio de Serra Negra, ante un auditorio compuesto por unos 100 investigadores, Beatriz Mendes, del Centro de Energía Nuclear en Agricultura (Cena), y Francisco Alves Mourão Filho, de la Escuela Superior de Agricultura Luiz de Queiroz (Esalq), ambos de la Universidad de São Paulo (USP), presentaron la primera variedad de naranja dulce genéticamente modificada, obtenida a partirde un tejido adulto.
Una conquista decisiva
En el año 2000, Mendes y Mourão ya habían obtenido una planta transgénica a partir de tejido cítrico joven, es decir, obtenido inmediatamente después de la germinación de la semilla, pero no quedaron satisfechos con ese resultado: la planta puede demorar entre cinco y ocho años en dar frutos. Trabajaron durante un año y medio analizando los factores que influyen sobre las condiciones de crecimiento de la planta – como el medio de cultivo, el tiempo de incubación y la temperatura. Finalmente, tuvieron éxito con el naranjo de la variedad Hamlin, a partir de su tejido adulto, que fructifica más rápidamente: en alrededor de dos años.
Es un logro que puede ser decisivo en la próxima etapa del proyecto, cuya conclusión está prevista para mediados de este año. “Cuando obtengamos un gen que le proporcione a la planta resistencia contra la bacteria, podremos producir plantas para pruebas de campo en unos dos años”, calcula Beatriz. Y otras investigaciones llegarán a su madurez este año. En la línea de frente de los resultados se encuentra João Lúcio de Azevedo, coordinador del Núcleo Integrado de Biotecnología (NIB) de la Universidad de Mogi das Cruzes (UMC) y profesor titular jubilado de la Esalq.
Azevedo desarrolla una Xylella mutante en la cual podrá ser bloqueado el gen de la enzima endoglucanasa A, uno de los genes asociados a la producción de la goma fastidiana. La bacteria utiliza esa goma para adherirse al xilema, el sistema de vasos que transporta agua y sales minerales a través de toda la planta.
La goma fastidiana está relacionada también con la obstrucción del xilema y, en consecuencia, a la manifestación de los síntomas de la enfermedad – manchas amarillentas – en las hojas del naranjo. Azevedo espera obtener durante 2002 los primeros resultados de la inoculación de la bacteria modificada en la planta vincapervinca rosa (Catharantus roseus), utilizada como modelo en este tipo de experimento, así como en los cítricos. “Si funciona”, dice Azevedo, “podremos hacer extensiva esta técnica a las bacterias del género Xantomonas, que atacan a los cítricos y a las hortalizas”.
Para avanzar, no se cuenta tan solo con informaciones acerca de cómo dicha bacteria provoca la enfermedad y la agresividad con la que la planta es infectada. Estos datos ya han sido profundizados con el conocimiento acumulado sobre el genoma de la Xylella, cuyo secuenciamiento contó con casi todos los investigadores que ahora participan en el proyecto Genoma Funcional. También se ha avanzado bastante en el conocimiento de las proteínas producidas por la causante de la plaga amarilla – el Laboratorio de Química de Proteínas del Instituto de Biología de la Universidad Estadual de Campinas (Unicamp) ya ha identificado 130 proteínas – y, de una manera más amplia, en la epidemiología de la enfermedad.
Experimentos llevados adelante principalmente en la Esalq muestran que, tanto la plaga amarilla cuanto la cigarrita, se propagan con mayor intensidad en las regiones más cálidas del estado de São Paulo, en donde la escasez de agua es frecuente. De esta manera, en el área de los municipios de Barretos y Bebedouro, en el norte del estado, un 48% de los naranjos está infectado, mientras que en los alrededores de Limeira e Itapetininga, en el sur, éstos no superan el 17%, de acuerdo con un estudio realizado en el año 2001 por el Fondo de Defensa de la Citricultura (Fundecitrus), solventado por los agricultores.
“Las etapas más importantes ya han sido transpuestas”, comenta Jesus Aparecido Ferro, de la Universidad Estadual Paulista (Unesp) de Jaboticabal, uno de los coordinadores del Genoma Funcional: “Con certeza, el progreso será más rápido de ahora en adelante”. Y si depende de la bióloga Patrícia Brant Monteiro, de la Fundecitrus, será aún más rápido: Monteiro produjo colonias de bacterias mutantes estables para 12 genes que regulan, entre otras cosas, la patogenicidad y la producción de toxinas para la planta o de polisacáridos (azúcares) que destruirán al xilema.
Tanto Patrícia como Azevedo trabajan en la técnica de la interrupción de genes. La “lectura” del gen determina la producción de proteínas específicas. Al colocar un tramo de ADN en el medio de un gen, dicha lectura se ve perturbada, y de ese modo, dicho gen es “desconectado”. La bacteria que resulta de esta modificación es una mutante, puesto que carga el gen alterado en su material genético.
Patricia fue pionera en la construcción de una Xylella mutante, resistente a la incorporación de material genético exótico – proveniente de otros organismos – a su genoma. La investigadora superó el problema al utilizar como vector un plásmido (segmento de ADN circular) desarrollado en laboratorio, que contenía un pequeño tramo de material genético de la propia bacteria. Este trabajo abrió el camino para otros grupos. Las investigadoras de la USP Marilis Marques, del Instituto de Ciencias Biomédicas, y Suely Gomes, del Instituto de Química, también tuvieron éxito en la producción de bacterias mutantes.
Utilizando una estrategia diferente, desarrollaron un plásmido que permitió la incorporación de ADN exótico al genoma de la Xylella. Marilis y Sueli desarrollaron colonias de Xylella que tienen el gen gspD alterado. Dicho gen es responsable por la producción de una proteína que forma canales en la pared de la bacteria, por donde son secretadas las enzimas que destruyen los vasos del xilema.
Recientemente, en pruebas realizadas utilizando material radiactivo para la marcación de genes, se comprobó que una de cada ocho colonias de bacterias mutantes mantiene incorporado el tramo de ADN alterado, aun después de diez ciclos reproductivos. “Comenzamos a dominar la técnica de transformación de la Xylella, y esto es importante para determinar la función de cada gen en la enfermedad”, revela Marilis. Ahora, la investigadora pretende producir bacterias mutantes utilizando transposones (pedazos de ADN que cambian de lugar en el cromosoma) como vectores, en lugar de usar plásmidos. La ventaja de esta técnica consistiría en obtener, a partir del uso de un único tipo de vector, varias colonias de bacterias transformadas, cada una con un gen diferente desconectado.
Investigaciones integradas
El dominio de la técnica de producción de mutantes fue igualmente esencial para el grupo coordinado en Piracicaba por Sérgio Pascholati, de la Esalq. Pascholati trabajó en la identificación de genes que codifican exoenzimas – proteínas que la bacteria produce y sirven para obtener nutrientes y colonizar a la planta. Valiéndose de las informaciones del genoma de la Xylella, identificó ocho posibles exoenzimas y, en testes de laboratorio, caracterizó tres de ellas: son tres celulasas, enzimas que digieren celulosa y la transforman en glucosa, una molécula esencial para que cualquier organismo obtenga energía. La próxima etapa consistirá en desarrollar bacterias con genes alterados que impidan la producción de esas proteínas.
Otro gen que los investigadores de Piracicaba pretenden desconectar es el Xf1940, productor de la enzima metionina sulfóxido reductasa. Esta enzima participa en el mecanismo de adhesión de la bacteria a la pared del xilema y a otras bacterias, para formar colonias, según el modelo desarrollado por Breno Leite, del equipo de Pascholati. La metionina también estaría relacionada con la fijación de la Xylella al aparato bucal de la cigarrita. Los investigadores confían en que podrán llegar a un mecanismo de control de la enfermedad bloqueando la acción de ese gen.
Pascholati trabaja en conjunto con especialistas del Fundecitrus, ligados también al equipo de Azevedo y al NIB de Mogi das Cruzes, que interactúa también con la Esalq y el Instituto Agronómico de Campinas (IAC). No se pretende llegar a una estrategia unificada para detener los estragos provocados por la plaga amarilla en la citricultura paulista. El propio equipo de Azevedo, más allá de bloquear genes, trabaja en otra forma de control de la plaga: por medio de microorganismos endofíticos, bacterias que conviven con la Xylella en los naranjos, pero que no provocan la enfermedad en la planta.
Su grupo identificó nueve géneros de bacterias endofíticas de cítricos. Entre ellas está la Pantoea agglomerans, en la cual los investigadores ya han logrado introducir un gen que produce xantanasa. Esta enzima impide la formación de la goma xantana, producida por las bacterias del género Xantomonas y similar a la goma fastidiana.
Objetivos seleccionados
“Tenemos que chequear todas las posibilidades”, dice el coordinador Ferro, de la Unesp: “No sabemos cuál de ellas servirá”. Patrícia pretende hacer pruebas con los mutantes en plantas durante este año, pero sabe que aún hay incertidumbre: “Será una suerte si conseguimos una Xylella no patogénica, pues las mitad de los genes de los organismos en general codifican proteínas de funciones aún desconocidas”.
Para que el combate contra la plaga sea eficiente, los científicos tuvieron que crear instrumentos que ayuden a determinar los mejores objetivos. Entre ellos está el microarreglo (microarray), también llamado “biochip“, una lámina de microscopio en donde son depositados los genes de la bacteria. El biochip indica, de una sola vez, cuáles son los genes – entre todos los del genoma – más activos en determinada situación.
Fue el grupo de Regina de Oliveira, del NIB de Mogi das Cruzes, que concluyó la tercera versión del biochip de la Xylella, con cerca de 2.500 genes – un 93% de los cerca de 2.700 que componen el material genético de la bacteria. “Es como si sacáramos una fotografía de la expresión génica de la célula en determinado momento”, explica Luiz Nunes, del NIB. Nunes ya ha identificado un conjunto de genes que la bacteria acciona, por ejemplo, como respuesta al estrés oxidativo – que se produce cuando es atacada por formas reactivas de oxígeno, como el peróxido de oxígeno, liberadas por el sistema de defensa de la planta hospedadora.
Regina y Nunes trabajan en cooperación con Sílvio Lopes, de la Unidad de Biotecnología de la Universidad de Ribeirão Preto(Unaerp), que estudia laactividad de los genes de cepas de laXylella que infectan a diferentes plantas. “Ya hemos identificado varios genes que creemos que están relacionados a la patogenicidad de la bacteria y a su especificidad con relación al hospedador”, dice Nunes.
Marcos Antônio Machado, del Centro de Citricultura del IAC, utilizó el biochip para comparar la expresión génica de la bacteria en dos situaciones de crecimiento: la de aislamiento primario, inmediatamente después de retirada de la planta, y la de los cultivos sucesivos, después de 25 ciclos de reproducción en laboratorio. En el primer estado, la Xylella se desarrolla lentamente en cultivo artificial, pero cuando es inoculada, coloniza a la planta rápidamente. En el segundo caso, ocurre lo contrario.
Banco de proteínas
La comparación entre ambas situaciones ha mostrado que algunos genes, vinculados a la capacidad de adhesión, se tornan menos activos cuando la Xylella es cultivada fuera de la planta. “Estos resultados comprueban que la capacidad de colonización está asociada a la de agregación”, dice Machado. “Tal vez podamos desarrollar alguna forma de reducir la acción de esos genes”. Para determinar cuáles son los genes más activos en la bacteria en una determinada situación, los especialistas del Laboratorio de Química de Proteínas del Instituto de Biología de la Unicamp adoptaron un abordaje diferente. En vez de analizar los genes directamente, observan el resultado final: las proteínas.
En cuatro meses, este grupo de Campinas, único en estudiar el Proteoma (el conjunto de las proteínas) de la Xylella, coordinado por José Camillo Novello, identificó 130 proteínas producidas por la bacteria. Las principales están asociadas a los procesos de adhesión y agregación, a la captación y al almacenamiento de hierro y a la eliminación de toxinas. También fueron encontradas proteínas de membrana, que actúan en la captación de nutrientes. Al final de la actual etapa del Genoma Funcional, prevista para su término a mediados de este año, el equipo de la Unicamp espera formar un banco de datos con entre 250 y 300 proteínas caracterizadas.
Medio de cultivo
El encuentro de Serra Negra significó también la superación de una de las etapas más complicadas del Genoma Funcional: el desarrollo de un medio de cultivo definido, en el cual se conocen todos los nutrientes – vitaminas, minerales, hidrocarburos y aminoácidos – necesarios para el crecimiento de la bacteria. Al cabo de dos años de trabajo, las investigadoras Eliana de Macedo Lemos y Lúcia Carareto Alves, de la Facultad de Ciencias Agrarias y Veterinarias de la Unesp de Jaboticabal, produjeron un medio de cultivo mínimo, que tiene como única fuente de nitrógeno al ácido aspártico (C4H7NO4).
“El estudio de las vías metabólicas demostró que la bacteria puede crecer en un medio relativamente sencillo, en condiciones muy similares a las del xilema”, dice Eliana. La determinación del medio de cultivo constituye una importante herramienta para aquéllos que trabajan con fisiología o genética de la Xylella, pues permite conocer genes expresados en determinadas condiciones y sirve de auxilio para la selección de formas mutantes de la bacteria. Y la Xylella que se cuide.
Republicar