En mayo de 2023 surgió una noticia prometedora en la lucha contra las bacterias resistentes a los medicamentos. Con la ayuda de la inteligencia artificial, científicos de Canadá y Estados Unidos identificaron un compuesto que presenta una acción potente y específica contra Acinetobacter baumannii, una bacteria que suele causar infecciones graves en los pulmones y en el tracto urinario de los pacientes internados en los hospitales. El referido patógeno aparece en la lista de bacterias elaborada en 2017 por la Organización Mundial de la Salud (OMS) contra las que urge buscar nuevos tratamientos pues los antibióticos actuales ya no logran eliminarlas.
El equipo dirigido por James Collins, del Instituto de Tecnología de Massachusetts (MIT), en Estados Unidos, y Jonathan Stokes, de la Universidad McMaster, en Canadá, entrenaron a un modelo informático para que reconozca las propiedades de 480 compuestos con alguna acción contra A. baumannii entre los 7.684 que ya se probaron para combatir a este microorganismo. Luego le presentaron al modelo otras 6.680 moléculas sin acción antibiótica conocida para que el programa detectara a aquellas con mayores probabilidades de matar al patógeno. En pocas horas, la lista de 6.680 compuestos se redujo a 240, algo que llevaría días o meses siguiendo los métodos de testeo tradicionales. Cuarenta de las 240 moléculas fueron capaces de inhibir el crecimiento de A. baumannii en laboratorio. Una en particular ‒ la denominada abaucina ‒ arrojó buenos resultados y pasó a probarse en animales. Aún queda un largo camino por recorrerse antes de que pueda convertirse en un antibiótico de uso humano.
Aunque el modelado computacional y la inteligencia artificial abrevian el tiempo invertido en el cribado de compuestos, no resolverán por sí solos la cuestión de la resistencia a los antibióticos. El problema es tan antiguo como el uso de estos compuestos en la medicina moderna y, a juicio de los expertos, requerirá diversas medidas: desde el uso estrictamente controlado de estos fármacos en la salud humana y animal hasta la prevención de infecciones mediante la higiene y, siempre que sea posible, la vacunación, además, claro está, del desarrollo de nuevos medicamentos.
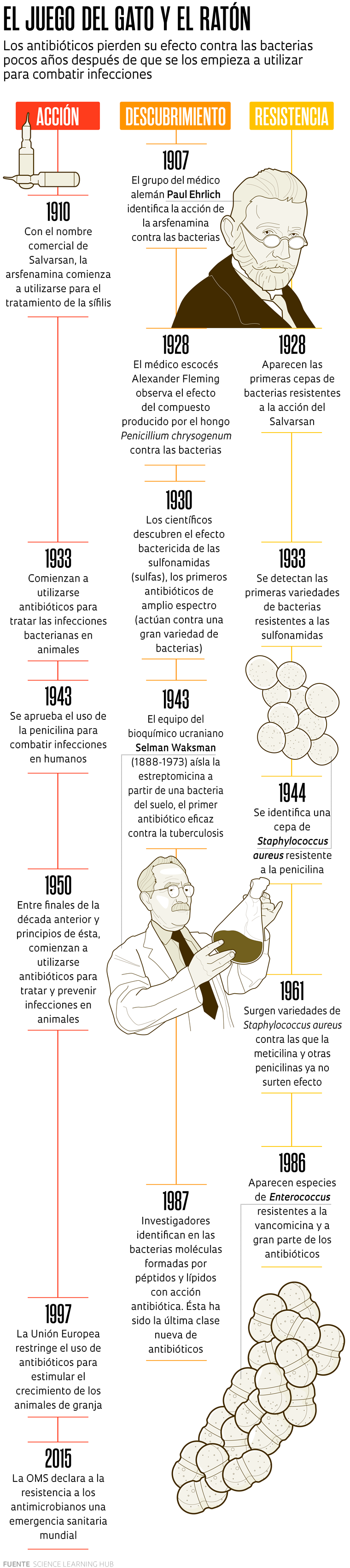
El primer antibiótico sintético conocido fue la arsfenamina, un compuesto que contiene arsénico identificado en 1907 en el laboratorio del médico alemán Paul Ehrlich (1854-1915), aunque existen indicios de preparados con productos naturales que ya se utilizaban en la Antigüedad.
En pruebas realizadas con conejos en 1909, Ehrlich y el bacteriólogo japonés Sahachiro Hata (1873-1938) demostraron que la arsfenamina era capaz de eliminar a la bacteria causante de la sífilis (Treponema pallidum) sin matar a esos animales. El estudio allanó el terreno para que, al año siguiente, el compuesto, bautizado con el nombre comercial de Salvarsan, pasara a utilizarse para el tratamiento de esta enfermedad de transmisión sexual que afectaba a la humanidad desde hacía siglos. Sin embargo, en 1928 aparecieron los primeros informes de casos en los que el Salvarsan ya no surtía efecto contra algunas variedades de T. pallidum.
Con la penicilina sucedió lo mismo. Este compuesto fue identificado en 1928 por el médico escocés Alexander Fleming (1881-1955) en un cultivo de la bacteria Staphylococcus aureus contaminado de manera casual con un hongo del género Penicillium en su laboratorio del Hospital Saint Mary’s de Londres, e inmediatamente empezó a utilizarse para el tratamiento de infecciones. Pasaría más de una década hasta que otros investigadores consiguieran purificar el principio activo y dominar su producción a gran escala. Sin embargo, incluso antes de que se generalizara el uso de la penicilina durante la Segunda Guerra Mundial, cuando salvó la vida de miles de soldados, ya había indicios de que las bacterias podían volverse resistentes al medicamento.
Fleming lo sabía y empezó a alertar sobre este problema, incluso en su discurso al recibir el Premio Nobel de Medicina en 1945. “No es difícil hacer que los microbios se vuelvan resistentes a la penicilina en el laboratorio, exponiéndolos a concentraciones insuficientes para matarlos. Existe el peligro de que alguien que lo ignore pueda fácilmente subdosificarse y, al exponer a sus microbios a cantidades no letales del fármaco, los haga resistentes”, dijo el médico escocés.
Y eso es lo que ha sucedido. El uso generalizado de estos compuestos en la salud humana, animal y en la producción agrícola ha sido acompañado por el surgimiento de bacterias resistentes a distintos antimicrobianos. “Las predicciones de Fleming resultaron ser certeras: la utilización incorrecta y, a veces, el abuso flagrante de antibióticos, acelera el desarrollo y la propagación de bacterias resistentes a ellos”, escribieron el químico Marco Terreni y sus colaboradores, de la Universidad de Pavía (Italia), en un artículo de revisión sobre nuevos antibióticos publicado en 2021 en la revista Molecules.

USDA-ARSAndrew Moyer (1899-1959), quien descubrió cómo producir penicilina a gran escalaUSDA-ARS
“Se asemeja a una partida de ajedrez”, dice la farmacéutica y bioquímica Ilana Camargo, coordinadora del Laboratorio de Epidemiología y Microbiología Molecular (LEMiMo), instalado en el campus de São Carlos de la Universidad de São Paulo (USP). Su grupo se dedica a caracterizar bacterias resistentes a múltiples antibióticos y a identificar nuevos compuestos con potencial para combatirlas. El último que han encontrado es un derivado sintético de la plantaricina 149, un péptido producido por la bacteria Lactobacillus plantarum. En las pruebas in vitro, esta molécula eliminó a 60 bacterias de diferentes especies y linajes con distintos grados de resistencia a los medicamentos disponibles en el mercado, según consta en un artículo publicado en febrero de 2023 en la revista Antibiotics. “Siempre que creemos que hemos puesto ‘en jaque’ a una bacteria con un nuevo antibiótico, esta encuentra enseguida una forma de burlarlo”, comenta.
Desde principios del siglo XX se han identificado decenas de antibióticos. Se trata de moléculas pertenecientes a distintos tipos que presentan diferentes mecanismos de acción. Un porcentaje importante de ellas ‒superior a un 75 %, según algunos investigadores‒ es de origen natural, producidas por otros microorganismos. Durante un largo período, entre las campeonas del suministro de nuevos antibióticos se encontraban las bacterias del género Streptomyces. Un artículo de revisión publicado en 2019 por investigadores del Reino Unido en la revista Current Opinion in Microbiology estima que dichas bacterias, presentes en el suelo y en la vegetación en descomposición, fueron la fuente de un 55 % de los antibióticos descubiertos entre 1945 y 1978. Entre ellos figuran la neomicina, la estreptomicina, la grisemicina y el cloranfenicol o cloromicetina. Empero, casi siempre, poco tiempo después de la introducción de un nuevo compuesto en el mercado, las bacterias se vuelven resistentes al mismo, y en algunos casos, a más de un antibiótico.
Hasta finales de la década de 1970, las grandes empresas de la industria farmacéutica lo solucionaban con el lanzamiento de un nuevo antibiótico, elaborado a partir de moléculas extraídas de otras bacterias u hongos y con una estructura química diferente a la de los anteriores. Pero estos compuestos empezaron a escasear porque se volvió más difícil hallarlos en los microorganismos más estudiados. “Los frutos al alcance de la mano ya habían sido recogidos”, relata Camargo, miembro del Centro de Investigación e Innovación en Biodiversidad y Fármacos (CIBFar), uno de los Centros de Investigación, Innovación y Difusión (Cepid), apoyados por la FAPESP. “Se hizo necesario invertir en investigaciones para modificar la estructura química de las moléculas que ya se conocían”, explica.
La facilidad con la que antes se obtenían nuevos antibióticos y el buen desempeño de los disponibles hasta entonces crearon una falsa sensación de seguridad, a lo que se sumó un cambio importante en la industria farmacéutica. A principios de los años 1980, una nueva generación de administradores asumió la dirección de las grandes empresas y reorientó el foco de las inversiones hacia el desarrollo de medicamentos más caros y rentables, destinados al tratamiento del cáncer y enfermedades asociadas al estilo de vida, como la diabetes, relatan el químico Matthew Todd y colaboradores, del University College de Londres, en el Reino Unido, en una publicación en 2021 de la revista Wellcome Open Research. “El problema central del pipeline de antibióticos vacío no es científico, sino económico”, dijeron.
Las utilidades obtenidas por la venta de los nuevos fármacos, más costosos y difíciles de obtener, por lo general no compensaban el costo de su desarrollo. Y según algunos especialistas, los ingresos por ventas caían aún más con el lanzamiento de las versiones genéricas de esos medicamentos. “Durante este período, varias grandes empresas cerraron departamentos enteros y discontinuaron sus programas de selección y desarrollo de antibióticos”, recuerda la bioquímica brasileña Andréa Dessen, coordinadora del Grupo de Patogénesis Bacteriana del Instituto de Biología Estructural (IBS), en Grenoble (Francia).