
Fundecitrus
Mapa genómico de los 2,7 millones de pares de bases del cromosoma de la X. fastidiosaFundecitrusDurante la mañana del día 1º de mayo de 1997, el físico José Fernando Perez, entonces director científico de la FAPESP, recibió un llamado telefónico del biólogo Fernando Reinach, en ese entonces profesor del Instituto de Química de la Universidad de São Paulo (IQ-USP). Desde el año anterior, ellos discutían nuevas formas de invertir en la calificación de los científicos brasileños en el área de genética molecular, considerada central para el dominio de la biotecnología. De esa larga conversación entre ambos nació aquello que se convirtió en el primer proyecto de secuenciación de un genoma en Brasil, el de la Xylella fastidiosa, la bacteria causante de la clorosis variegada de los cítricos (CVC), una de las peores plagas de los naranjales de São Paulo. Lanzado el 14 de octubre de ese año, el proyecto abarcó a 35 laboratorios y a 191 investigadores de diversas instituciones del estado y de diferentes disciplinas, integrados virtualmente por la Red de Organización para la Secuenciación y el Análisis de Nucleótidos (Onsa, en inglés). Además de inaugurar la investigación en genética molecular, la primera secuenciación de un fitopatógeno de plantas en el mundo constituyó una nueva forma de hacer ciencia en Brasil, y contribuyó a la formación y la calificación de una generación de jóvenes científicos. Muchos se valieron del conocimiento y la experiencia adquiridos para abrir sus propias empresas. Otros cambiaron el enfoque de sus investigaciones, formaron nuevas redes de colaboración científica y avanzaron en sus carreras dentro y fuera de la universidad.
El mapeo del genoma de la Xylella amplió las perspectivas de investigación en genética en Brasil. Otras secuenciaciones se han concretado a partir de entonces, como el proyecto Genoma Caña, que concluyó en noviembre de 2000, con el objetivo de secuenciar partes seleccionadas del ADN de la caña de azúcar e identificar genes con características de interés económico. Casi al mismo tiempo surgieron los proyectos Genoma Cáncer, finalizado en marzo de 2002, y de secuenciación del código genético de la bacteria Xanthomonas citri, causante del cancro cítrico, concluido en agosto de 2001. Esos emprendimientos científicos ayudaron a capacitar a los investigadores y a mejorar la infraestructura de los principales laboratorios de São Paulo, acelerando el desarrollo de la biología molecular en Brasil.

Portada de la edición de la revista Nature, que consideró a la secuenciación del fitopatógeno un hito en la biociencia brasileña
La experiencia adquirida al participar en el proyecto y el prometedor mercado de productos y soluciones biotecnológicas también incentivaron a varios investigadores a lanzarse como emprendedores. En 2002, dos años después de la conclusión de la secuenciación, el científico de la computación João Paulo Kitajima dejó el Laboratorio de Bioinformática (LBI) en el Instituto de Computación de la Universidad de Campinas (IC-Unicamp), donde trabajaba, para fundar, junto con otros integrantes del proyecto, Alellyx, una empresa de investigación y desarrollo de productos de biotecnología orientada a la generación y comercialización de patentes en genómica aplicada (lea en Pesquisa FAPESP, edición nº 74).
Kitajima se unió al equipo de bioinformática inmediatamente después del inicio del proyecto genoma de la Xylella. “Empecé como analista de bioinformática en el LBI al lado de los bioinformáticos João Meidanis y João Carlos Setubal. Y pasé a ayudar en la coordinación de la parte de bioinformática del proyecto: era el tercer ‘João’ del liderazgo”, comenta. En esa época, frente a la necesidad de que hubiera una comunicación rápida entre los laboratorios implicados, la dirección del proyecto optó por la creación de una red virtual, con una coordinación central, físicamente distribuida por los centros de investigación del estado de São Paulo. Lanzada en diciembre de 1997, la red Onsa interconectó los laboratorios, estableciendo vínculos entre investigadores y posibilitando el desarrollo de protocolos genéticos, el intercambio de información, la resolución de problemas y la adaptación y el ajuste de técnicas.

Fundecitrus
El insecto transmisor de la bacteriaFundecitrusLa red contaba con un sistema de bioinformática para que los investigadores pudieran incorporar la información resultante de la secuenciación del material genético de la bacteria. Les cupo a Setubal, Meidanis y Kitajima garantizar el mantenimiento del flujo de información. “Nos encargábamos de recibir los datos de secuenciación producidos por los laboratorios y procesarlos computacionalmente de manera tal de generar un genoma completo”, comenta Setúbal. “No sabíamos si los grupos tendrían acceso a una banda de internet suficiente como para enviarnos todas la información genómica”, recuerda Meidanis, quien entró en contacto con la bioinformática en su doctorado en la Universidad de Wisconsin-Madison, en Estados Unidos, en 1989.
“En Alellyx tuve la oportunidad de coordinar la investigación aplicada y de especializarme en el área de procesos regulatorios de organismos transgénicos”, comenta el biólogo Jesus Aparecido Ferro, de la Facultad de Ciencias Agrarias y Veterinarias de la Universidade Estadual Paulista (Unesp) en Jaboticabal y uno de los fundadores de la empresa. Ferro también fue uno de los coordinadores del programa Genoma Funcional de la Xylella, lanzado en junio de 1999. El proyecto tenía el propósito de investigar la función de los genes identificados a lo largo de la secuenciación, a los efectos de comprender de qué manera la Xylella desencadenaba la CVC.

Eduardo Cesar
Ana Claúdia Rasera, João Kitajima, Jesus Ferro, Paulo Arruda y João Setúbal durante el lanzamiento de la empresa de biotecnología Alellyx, en 2002Eduardo CesarEn 2008, Alellyx fue vendida junto con la empresa CanaVialis a la multinacional Monsanto por un valor de 290 millones de dólares (el equivalente actual a 980 millones de reales). “En 2009 fui al área de salud humana”, comenta Kitajima. “Trabajé como bioinformático en el Instituto Israelita de Enseñanza e Investigación del Hospital Albert Einstein hasta 2011”. Recientemente, él y otros socios fundaron Mendelics Análise Genômica, una empresa de diagnósticos genómicos personalizados.
Para Meidanis, el proyecto implicó un gran cambio en su vida. “Me convertí en un híbrido entre académico y director de empresa”, dice. Actualmente reparte su tiempo entre el cargo de profesor en la Unicamp y el de empresario en Scylla Bioinformática, la empresa de software para el desarrollo de la genómica y la proteómica que fundó.

Adri Felden/ Ag. Argos
La ceremonia de entrega de la Medalla Paulista al Mérito Científico y Tecnológico…Adri Felden/ Ag. ArgosUn proyecto ambicioso
El proyecto genoma de la Xylella se puso en marcha durante un período de fuerte incidencia de investigaciones en el campo de la biología molecular. Sin embargo, esa ciencia estaba en mantillas en Brasil. “Perez se percató de que la producción científica en ese campo crecía en otros países y que era necesario calificar a los investigadores brasileños para que pudiésemos ascender a una posición de mayor relieve en el escenario científico internacional”, comenta Reinach en su oficina en el Fondo Pitanga, un agrupamiento de recursos de inversores interesados en emprendimientos con base tecnológica. A partir de entonces, se dio inicio a una larga articulación científica alrededor de la elaboración del proyecto. “Había desconfianza por parte de los investigadores más antiguos”, comenta. “Se trataba de un proyecto ambicioso, arriesgado y que requeriría mucho dinero”, dice el biólogo, quien en esa época era uno de los pocos científicos brasileños con experiencia en el campo de la biología molecular y de los emprendimientos científicos, habiendo sido fundador de la primera empresa de test de paternidad en el país, Genomic Engenharia Molecular.
La elección del microorganismo que sería secuenciado se produjo semanas antes del lanzamiento del proyecto. “El genoma debía ser lo suficientemente grande como para involucrar a muchos investigadores y lo suficientemente pequeño como para que lográsemos concluirlo”, comenta Perez, en su despacho en Recepta Biopharma, la empresa de biotecnología que creó en 2005, orientada a la investigación y el desarrollo de compuestos con potencial para combatir el cáncer.

Adri Felden/ Ag. Argos
…, otorgada por la gobernación del estado de São Paulo a los 191 investigadores que tomaron parte en el proyecto genomaAdri Felden/ Ag. ArgosEn aquel momento, el organismo que mejor cumplía con esos requisitos previos era el Thiobacillus ferrooxidans, utilizado en biominería. A su vez, la Xylella era una bacteria que ningún grupo brasileño había logrado cultivar. “En septiembre de 1997, el Fondo de Defensa de la Citricultura [Fundecitrus] manifestó su interés por participar en el proyecto en caso de que la Xylella fuese seleccionada”, comenta Perez. “Mencioné las dificultades que revestía la elección de la bacteria, pero ellos me rebatieron diciendo que un investigador francés sabía cómo cultivarla”. Se referían a Joseph Bové, del Instituto Nacional de Investigación Agronómica (Inra, en francés), en Burdeos, Francia, que trabajaba con genética molecular y que se había dispuesto a suministrar la bacteria, mientras que Frédéric Laigret, de la Universidad Burdeos II, daría apoyo a la constitución de un banco de ADN de la bacteria para el suministro de los fragmentos que serían secuenciados. “Fueron convincentes y elegimos a la Xylella”, dice Perez.
El proyecto se lanzó con una inversión de 12 millones de dólares de la FAPESP y otros 400 mil dólares por parte de Fundecitrus, el mayor valor hasta entonces destinado a un emprendimiento científico en Brasil. De los 70 laboratorios que se postularon para participar del proyecto, 35 fueron seleccionados por un comité internacional formado por Steve Oliver, de la Universidad de Manchester, Inglaterra, André Goffeau, de la Universidad Católica de Lovaina, Bélgica, y John Sgouros, del Imperial Cancer Research Foundation, Inglaterra. Ese mismo comité se encargó a su vez de supervisar la marcha del proyecto.

Adri Felden/ Ag. Argos
El exgobernador de São Paulo Mario Covas condecora al bioquímico Andrew Simpson (a la izq.) por su liderazgo en la coordinación del proyectoAdri Felden/ Ag. ArgosHabida cuenta de la inexperiencia de los investigadores brasileños en el campo de la biología molecular, se decidió que el proyecto contaría con coordinadores de ADN y bioinformática, además de laboratorios centrales de secuenciación para capacitar a los investigadores y generar las secuencias necesarias. La coordinación de ADN permaneció a cargo del bioquímico Andrew Simpson, del Instituto Ludwig de Investigaciones sobre el Cáncer, en São Paulo. Los laboratorios centrales de secuenciación fueron el Laboratorio de Biología Molecular del IQ-USP, en esa época encabezado por Reinach, y el Centro de Biología Molecular e Ingeniería Genética (CBMEG) de la Unicamp, coordinado por el biólogo Paulo Arruda. Los laboratorios seleccionados recibieron secuenciadores automáticos de ADN, reactivos y asesoría técnica. La Xylella sería mapeada mediante 99 cósmidos, fragmentos de ADN que cargan y multiplican partes del genoma que se pretende estudiar.
Menos de un año después del lanzamiento del proyecto, el 90% del código genético de la Xylella había sido secuenciado. Restaban aún algunos agujeros, o interrupciones de la secuencia total del genoma, llamados gaps. Para cerrarlos y confirmar el orden de los cósmidos, Simpson desarrolló abordajes experimentales alternativos en los laboratorios del Instituto Ludwig, mientras que intentaba unir esos cósmidos a los contiguos, de modo tal de ordenar las secuencias y describir completamente el genoma.

Gilberto Alves
El grupo de científicos responsable de la secuenciación de la Xylella es recibido por el expresidente de Brasil, Fernando Henrique Cardoso (de traje claro), en el Palacio de AlvoradaGilberto AlvesNuevas oportunidades
La genetista Anamaria Camargo se sumó al equipo encabezado por Simpson en el Instituto Ludwig en 1998, en su pasantía posdoctoral. Su trabajo consistió en el cierre de los gaps del genoma de la Xylella junto a las investigadoras Cláudia Monteiro-Vitorello, Elizabeth Martins, Mariana Cabral de Oliveira, Marie-Anne Van Sluys, Marilis do Valle Marques y Ana Cláudia Rasera. “Hoy en día trabajo en el área de genómica del cáncer utilizando mucho de lo que aprendí en el proyecto de la Xylella”, comenta Camargo, coordinadora del Centro de Oncología Molecular del Instituto Sirio-Libanés de Enseñanza e Investigación, en São Paulo. Su colega, la bióloga Marilis do Valle Marques, se involucró en el proyecto por invitación de la física Suely Lopes Gomes, docente del IQ-USP. “Participé en el análisis y el montaje de las secuencias y del montaje y la anotación del plásmido pXF51, lo que constituyó un desafío, pues tenía una región duplicada que dificultaba el montaje de la secuencia”, explica Do Valle Marques, docente del Departamento de Microbiología del Instituto de Ciencias Biomédicas (ICB) de la USP.
El proyecto quedó concluido en enero de 2000 y consistió en el secuenciación de 2,7 millones de pares de bases del cromosoma de la Xylella. El día 13 de julio de ese año, Nature le dedicó la portada de la revista al trabajo. En Brasil, los científicos recibieron premios y homenajes, como la Medalla Paulista al Mérito Científico y Tecnológico, concedida por la gobernación del estado de São Paulo.

Eduardo Cesar | Léo Ramos Chaves
Van Sluys y Cabral de Oliveira: experiencia adquirida en Brasil y liderazgo en proyectos en Estados UnidosEduardo Cesar | Léo Ramos ChavesPara Simpson, el éxito del proyecto genoma de la Xylella le permitió encabezar otros proyectos, como el del Genoma del Cáncer. “Como resultado de esos trabajos, ascendí al puesto de director científico del Instituto Ludwig en Nueva York”, comenta. Años después regresó a Brasil como director presidente de la empresa de biotecnología Orygen, que trabaja en el desarrollo, la fabricación y comercialización de anticuerpos terapéuticos y vacunas.
El proyecto genoma de la Xylella se transformó en una oportunidad para que muchos jóvenes científicos se consolidasen como investigadores independientes. Tras participar en el proyecto, el biólogo Marcelo Briones, del Departamento de Microbiología de la Universidad Federal de São Paulo (Unifesp), se convirtió en uno de los coordinadores del proyecto Genoma Cáncer junto a Simpson. En ese entonces, los equipos usados en su laboratorio para la secuenciación de los cósmidos de la Xylella lo ayudaron a secuenciar genes expresados en cánceres de gran incidencia en Brasil, tales como los de cuello uterino y de cabeza y cuello. En 2000, tras la conclusión de ambos proyectos, Briones recibió una financiación del Instituto de Medicina Howard Hughes, en Estados Unidos, para desarrollar investigaciones sobre enfermedades infecciosas y parasitarias en Brasil. “Sin los equipos y el conocimiento adquirido durante los proyectos genoma de la Xylella y del Cáncer, no habría obtenido la financiación”, comenta.

Márcia Minilo
Perez y Reinach: un enfoque en las nuevas formas de calificar a los investigadores brasileñosMárcia Minilo“Toda mi carrera se basa en el estudio de la Xylella”, dice la bióloga Alessandra Alves de Souza, del Centro de Citricultura Sylvio Moreira, del Instituto Agronómico de Campinas (IAC), con sede en la ciudad de Cordeirópolis, interior paulista. Graduada en el estado de Pernambuco, Alves de Souza llegó a São Paulo cuando ingresó a la maestría en la Escuela Superior de Agricultura Luiz de Queiroz (Esalq) de la USP. En el doctorado, en la Unicamp, realizó análisis comparativos de la expresión de genes de la Xylella asociados con la patogenicidad y con la formación de una biopelícula que une a la comunidad de microorganismos invasores en las plantas. Esto la llevó a estudiar una molécula llamada N-acetilcisteína (NAC) como posible alternativa de control de la CVC. El NAC es un compuesto utilizado para desobstruir las vías respiratorias y reducir las biopelículas formados por bacterias que causan enfermedades en humanos. En pruebas preliminares, el NAC se mostró eficaz al romper las biopelículas producidos por la Xylella, disminuyendo los síntomas de la CVC. Recientemente, Alves de Souza y la bióloga Simone Picchi, también del IAC, abrieron CiaCamp, una empresa de investigación y desarrollo de fertilizantes a base de NAC (lea en Pesquisa FAPESP, edición nº 247). Alves de Souza participa actualmente en un proyecto financiado por la Comisión Europea para el combate de una variante de la Xylella de Italia, que está destruyendo los olivos de la región de Apulia.
La bióloga Cláudia Monteiro-Vitorello ya había trabajado con la secuenciación de ADN mitocondrial de un hongo causante de enfermedades en castaños norteamericanos durante su posdoctorado en la Universidad del Estado de Michigan, en Estados Unidos. Cuando regresó a Brasil, en 1998, la invitaron a integrar el equipo del ingeniero agrónomo Luiz Lehmann Coutinho, del Departamento de Ciencia Animal de la Esalq-USP. “Durante el proyecto de la Xylella participé en un curso en el Laboratorio Nacional de Computación Científica (LNCC), en Río de Janeiro”, comenta. Allí conoció a Amos Bairoch, coordinador del Swiss-Prot, un banco de datos de proteínas suizo. “Habida cuenta de ese encuentro y del proyecto de la Xylella, me contrataron para trabajar en el Swiss-Prot en la parte de anotaciones de genomas de bacterias”, recuerda. En 2008, Moneiro-Vitorello volvió a la universidad como profesora adjunta de Microbiología en la Universidad Federal del ABC, en Santo André. En 2009 se trasladó a la Esalq-USP, en donde trabaja en el área de genómica de la interacción planta-patógeno con enfermedades de la caña de azúcar.
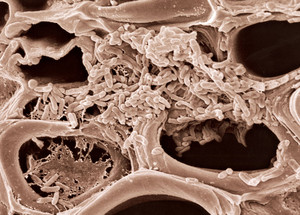
FRANCISCO TANAKAE ELLIOT KITAJIMA/ ESALQ-USP
Imagen de microscopía de las biopelículas formadas por la Xylella en el interior de las plantasFRANCISCO TANAKAE ELLIOT KITAJIMA/ ESALQ-USPPara la bióloga Marie-Anne Van Sluys, del Departamento de Botánica del Instituto de Biociencias (IB) de la USP, el proyecto genoma de la Xylella fue una oportunidad para transformar sus estudios sobre la interacción entre las bacterias y las plantas en el enfoque principal de sus investigaciones. Luego de terminar la secuenciación del genoma de la Xylella, Van Sluys y la bióloga Mariana Cabral de Oliveira, también del IB-USP, encabezaron un proyecto orientado a la decodificación de los genomas de una cepa de la Xylella responsable de la destrucción de las vides en Estados Unidos y de la bacteria Leifsonia xyli subsp xyli, que ataca el tallo de la caña de azúcar. “También participamos de una red liderada por el Joint Genome Institute, de Estados Unidos, para develar el ADN de otras dos cepas de la Xylella que acometen al almendro y que se instalan en una planta conocida como laurel de flor”, comenta.
“Fue una época de gran interacción con científicos de diversas áreas”, comenta Cabral de Oliveira. La bióloga participó en todas las etapas del proyecto del genoma de la Xylella, desde la secuenciación hasta el análisis macroestructural y comparativo de los genomas, lo que le permitió posteriormente transferir las técnicas de análisis de secuencias a gran escala a su estudio con algas. Para Perez, este proyecto demostró que Brasil es capaz de hacer investigación en forma colaborativa y abarcando a la comunidad científica, al poder público y a la iniciativa privada. “Y más que eso: le permitió al país cambiar su percepción sobre sus capacidades y su lugar en el mundo de la ciencia.”
Republicar
