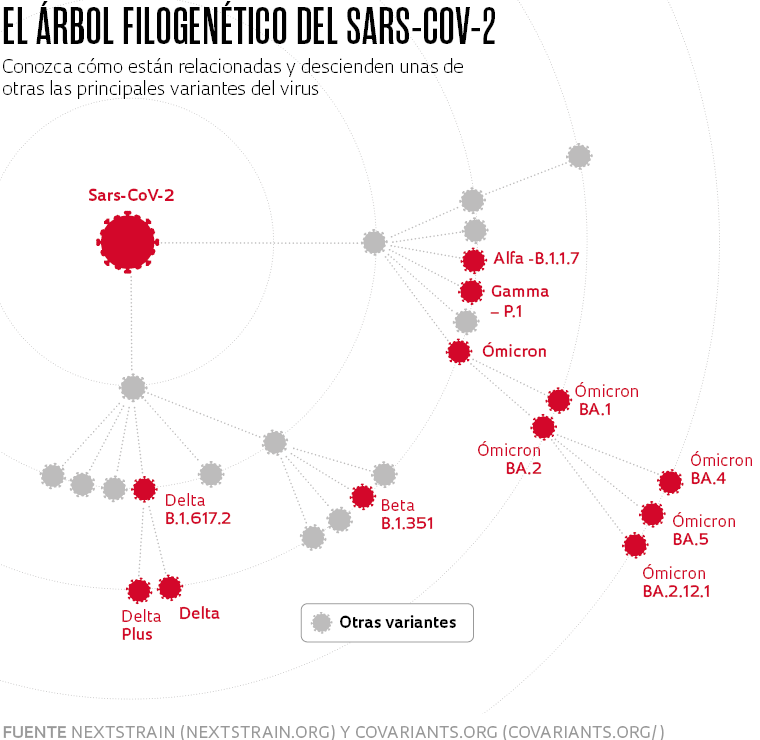
¿Cuáles son las variantes circulantes en Brasil del virus que causa el covid-19 y cuál es su impacto sobre la salud de la población? ¿Los nuevos linajes del Sars-CoV-2 se propagan con mayor facilidad? ¿Causan una enfermedad más o menos grave? ¿Las vacunas y los test de diagnóstico siguen siendo eficaces? Estas son algunas de las preguntas que pretende responder el trabajo de vigilancia genómica para saber cuáles son las mutaciones presentes en los virus en circulación. Los especialistas en esta materia son quienes darán el alerta cuando surja una nueva variante de preocupación –aquellas que suponen un mayor riesgo potencial para la salud pública–, ya sea por ser más transmisible o más letal.
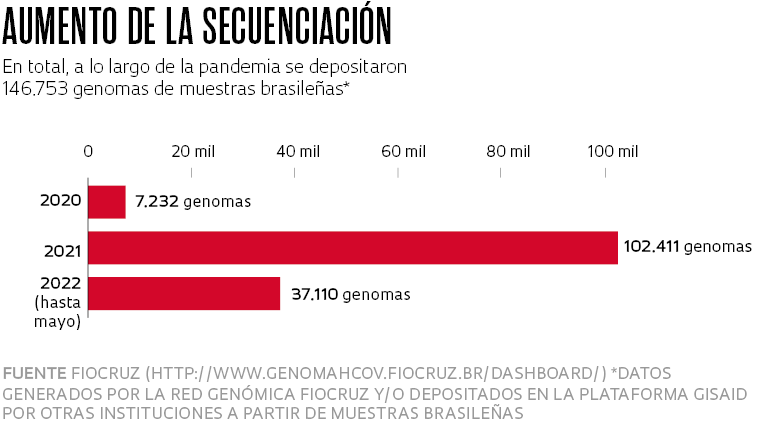
En Brasil, según la plataforma internacional Gisaid, solamente se han secuenciado muestras del 0,47 % de los más de 31 millones de casos registrados de la enfermedad y, por lo tanto, el material genético decodificado se ubica muy por debajo del 5 % recomendado por los expertos. En cifras concretas, se han analizado menos de 150.000 genomas. El porcentaje ha sido menor, principalmente en el primer año de la pandemia, debido a la falta de financiación, de equipos y de insumos. Gisaid es la principal base internacional de intercambio de datos sobre el código genético y las mutaciones detectadas en el nuevo coronavirus.
Aunque las cifras de la vigilancia genómica en el país han mejorado a partir del segundo semestre de 2021 (obsérvese la infografía), los investigadores y gestores siguen buscando recursos para garantizar el mantenimiento y la ampliación de los trabajos. La buena noticia es que las dos grandes redes nacionales de vigilancia genómica han conseguido recientemente el apoyo de la Agencia de Seguridad Sanitaria del Reino Unido (UKHSA) para intensificar los trabajos de secuenciación genética e identificar posibles nuevas variantes en circulación en el país.
Investigadores de la Red Genómica Fiocruz, que cuenta con el apoyo del Ministerio de Salud (MS) y cuya coordinación está a cargo de científicos de la Fundación Oswaldo Cruz (Fiocruz), y de la Red Nacional de Ómicas del Covid-19 (Red Corona-ómica BR), financiada por el Ministerio de Ciencia, Tecnología e Innovación (MCTI), han participado en reuniones de trabajo con miembros de la plataforma NVAP (New Variant Assessment Platform). El programa fue creado el año pasado por el gobierno británico para reforzar la infraestructura de otros países y colaborar en la detección de nuevas variantes. Además de Brasil, también forman parte de la iniciativa Chile, Singapur, Etiopía, Kenia, Nigeria, Pakistán y Trinidad y Tobago.
“La ayuda de la agencia gubernamental británica no es la única, pero representa un respaldo importante para proseguir con nuestros análisis y poder generar información genómica”, pone de relieve la viróloga Marilda Siqueira, jefa del Laboratorio de Virus Respiratorios y Sarampión del Instituto Oswaldo Cruz (IOC/Fiocruz). Esta unidad, un centro de investigación, desarrollo y capacitación en el área de los virus que afectan el sistema respiratorio, es una referencia en materia de covid-19 para el MS, en Brasil, y para la Organización Mundial de la Salud (OMS), en toda América.
Responsable a cargo de la Red Genómica Fiocruz, Siqueira dice que el apoyo británico hará posible que otras unidades de la Fiocruz comiencen a realizar análisis genómicos del Sars-CoV-2. Por ahora, tan solo ocho de los once centros de la institución llevan a cabo este tipo de trabajo en los estados de Amazonas, Ceará, Pernambuco, Piauí, Bahía, Río de Janeiro, Minas Gerais y Paraná.
“La realización del análisis genómico es una tarea costosa y compleja. No solo requiere personal calificado, sino equipamiento e insumos onerosos”, explica la investigadora. Según ella, la ayuda británica, cuya duración está prevista en un año, contempla, en el caso de la red Fiocruz, la provisión de insumos y capacitación del personal en las áreas de bioinformática, análisis de datos y vigilancia epidemiológica. “Empezamos a recibir los insumos en el mes de abril y ya hemos realizado algunas capacitaciones”.
En el otro frente de apoyo a Brasil, coordinado por la Red Corona-ómica BR, el acuerdo con los británicos se selló poco después y, hasta finales de junio, los insumos aún no habían llegado, aunque los expertos del Reino Unido ya estaban llevando a cabo workshops con el equipo de la red local. Dicha red, creada en 2020, agrupa a 19 núcleos de investigación, principalmente de universidades. “La idea de los británicos es apoyar a las redes ya establecidas y ceder los insumos para que podamos profundizar en las tareas de secuenciación genómica y vigilancia epidemiológica”, dice el virólogo Fernando Spilki, coordinador de la red y docente de la Universidad Feevale, en Nova Hamburgo (en Rio Grande do Sul).
“Para nosotros, lo más importante con relación a este proyecto tiene que ver con la formación de personal. El Reino Unido posee una gran experiencia en materia de cruzamiento de los datos de la vigilancia genómica con la información epidemiológica”, dice el investigador. “La determinación de las correlaciones entre una determinada variante o sublinaje y un brote o un incremento de los casos puede parecer trivial, pero para este análisis deben tenerse en cuenta muchas variables, entre las cuales se cuentan los aspectos sociales, geográficos, económicos y temporales”.
Uno de los proyectos en el marco de la Red Corona-ómica BR que obtuvieron el respaldo de la plataforma NVAP está a cargo de la médica Ester Sabino, docente del Instituto de Medicina Tropical (IMT) de la Facultad de Medicina de la Universidad de São Paulo (FM-USP), y coordinadora del Centro Conjunto Brasil-Reino Unido para el Descubrimiento, el Diagnóstico, la Genómica y la Epidemiología de los Arbovirus (Cadde), financiado por la FAPESP y por la agencia británica de financiación de la investigación en salud Medical Research Council (lea en Pesquisa FAPESP, edición nº 304).
En 2021, Sabino inició un proyecto de vigilancia genómica con el apoyo de la empresa minorista Magalu, en el marco de una iniciativa del Grupo de Mujeres de Brasil. La propuesta inicial consistía en la creación de un grupo en cada capital brasileña que trabajara en red y secuenciara 100 muestras del virus del covid-19 por mes.

Adenir Britto / PMSJCTest de detección de covid: la caída en la realización de exámenes moleculares RT-PCR dificulta las tareas de vigilancia genómicaAdenir Britto / PMSJC
“Los recursos llegaron en noviembre, contratamos a las coordinadoras, recibimos los primeros reactivos y comenzamos a capacitar al personal”, relata Sabino. La iniciativa ya se ha concretado en 19 capitales, incluso en estados alejados del centro y sur del país, como Amapá, Acre y Rondônia, que rara vez son incluidos en proyectos de gran porte porque carecen de infraestructura, informa la investigadora. Algunos laboratorios ya están realizando los análisis genómicos. El convenio inicial con el grupo empresario prevé provisión de recursos para seis meses de trabajo.
“Esta iniciativa del Grupo de Mujeres de Brasil es importante porque está capacitando a grupos que no tenían experiencia con la secuenciación”, comenta el virólogo Renato Santana, del Departamento de Genética, Ecología y Evolución del Instituto de Ciencias Biológicas de la Universidad Federal de Minas Gerais (ICB-UFMG), cuyo laboratorio forma parte del paraguas de la Red Corona-ómica. “Están recibiendo equipamiento e insumos para llevar a cabo la secuenciación y el seguimiento del Sars-CoV-2 en sus propios centros”.
El laboratorio que dirige Santana también recibirá reactivos en el contexto de la plataforma NVAP, al igual que los laboratorios contemplados en el proyecto de Mujeres de Brasil y otros de la Red Corona-ómica. “Tenemos previsto secuenciar al menos 5.000 muestras por mes en las cinco regiones de Brasil”, informa el investigador.
Un colaborador de peso para la ejecución del proyecto, comenta Santana, es el grupo Hermes Pardini, con sede en la ciudad de Belo Horizonte, cuya red cuenta con más de 6.000 laboratorios de análisis clínicos asociados en todo el país. “La totalidad de las 16.000 muestras positivas de covid-19 diagnosticadas por el grupo desde el mes de enero de este año ya se encuentran en mi laboratorio. Solo estamos aguardando la llegada de los reactivos para reenviarlos a los laboratorios que llevarán a cabo la secuenciación”, afirma.